










Preview text:
lOMoAR cPSD| 45469857
Phương pháp và kỹ thuật cơ bản trong sinh học phân tử I.
Các công cụ cơ bản
1. Chủng vi sinh vật
Các nghiên cứu SHPT thường tiến hành trên Ecoli Bộ
máy di truyền đã được nghiên cứu khá đầy đủ
Tốc độ tăng trưởng nhanh
Khả năng gây bệnh lại thấp
Đặc điểm một số chủng xuất phát từ Ecoli K12 JM109:
- dùng để nhân bản các vector của phage M13
- có thể dùng với plasmid pUC sử dụng kỹ thuật bổ sung alpha để
chọn lọc dòng tái tổ hợp
- kỹ thuật bổ sung alpha: chủng bị mất gen B-galactosidase trên
NST, nhưng có mang gen cho đoạn omega của B-galactosidase
trên plasmid F', cho phép nó bị nhiễm bởi các phage dạng sợi như M13
- Nếu mất plasmid F' sẽ làm mất khả năng này, do đó phải duy trì
chủng này trên moi trường tối thiểu
- Đột biến trên gen recA, làm mất khả năng tái tổ hợp tương đông
- chủng này được ưa dùng hơn JM103 nếu tái tổ hợp là các đoàn
chèn thay đổi. Tuy nhiên, hiệu suất thu ADN của M13DH5-α
- có đặc tính bổ sung alpha (giống JM103 và JM109) - đột biến recA1
- đột biến deoR làm => dễ dàng cho việc thu nhận các mảnh ADN lớn DH5-αF':
- rất tốt cho việc nhân bản vector M13 mp
- cho phép tạo ra ADN sợi đơn từ plasmid có origin f1 (như vector
M13) khi bị nhiễm đồng thơi một phage trợ giúp (helper phage) lOMoAR cPSD| 45469857
- khác với JM103, F' episome, cần để nhân bản phage sợi đơn, ổn
định, ko cần sử dụng mtr tối thiểu để duy trì nó 2. Vector tạo dòng
- Vật liệu di truyền trung gian để chuyển gen giữa các tb
- sao chép và phân bố vào tb mới trong quá trình phân bào
- gen nghiên cứu sao chép và hiện diện ở tất cả các tb xuất phát từ
tb nhận gen ban đầu - Yêu cầu •
Có thể được sao chép bởi tế bào nhận nó. •
Có trình tự nhận diện của enzym giới hạn để cắt và đưa gen ngoại lai vào. •
Mang một hay nhiều yếu tố chọn lọc nào đó để phân biệt hoặc
chọn lọc tế bào nhận được vector với tế bào không nhận được. Plasmid:
- Plasmid là một phân tử ADN sợi đôi, dạng vòng kín, nhỏ có thể
nhân bản độc lập với nhiễm sắc thể trong tế bào - yêu cầu: •
có yếu tố đánh dấu để chọn lọc dòng tái tổ hợp: 1 hay nhiều gen kháng sinh •
có điểm khởi đầu sao chép (Ori) cho phép nhân bản plasmid
trong tbc không phụ thuộc NST. Điểm Ori có thể kiểm soát
được , vd cho phép khuếch đại plasmid khi có mặt chất ức chế
tổng hợp protein (như chloramphenicol), tăng số bản sao của
plasmid trong tế bào chủ và cho hiệu suất cao khi chiết tách
plasmid với độ tinh khiết khá tốt. Trình tự của Ori cũng quyết
định số bản sao của plasmid trong một tế bào. •
Có vùng tạo dòng (Multiple Cloning Site - MCS): là một trình tự
ADN ngắn, mang nhiều trình tự nhận diện duy nhất của các RE
khác nhau nằm liên tiếp (polylinker), cho phép cắt để mở vòng
plasmid và nối đoạn gen mong muốn vào.Có yếu tố đánh dấu
để chọn lọc dòng tái tổ hợp, thường là một hay nhiều gen kháng kháng sinh.
- Plasmid có thể mang yếu tố đánh dấu chèn để nhận biết plasmid
sau khi đưa vào tế bào đã được nối với gen ngoại lai trong vùng tạo dòng hay chưa.
- Được đưa vào tế bào chủ bằng cách biến nạp (transformation)
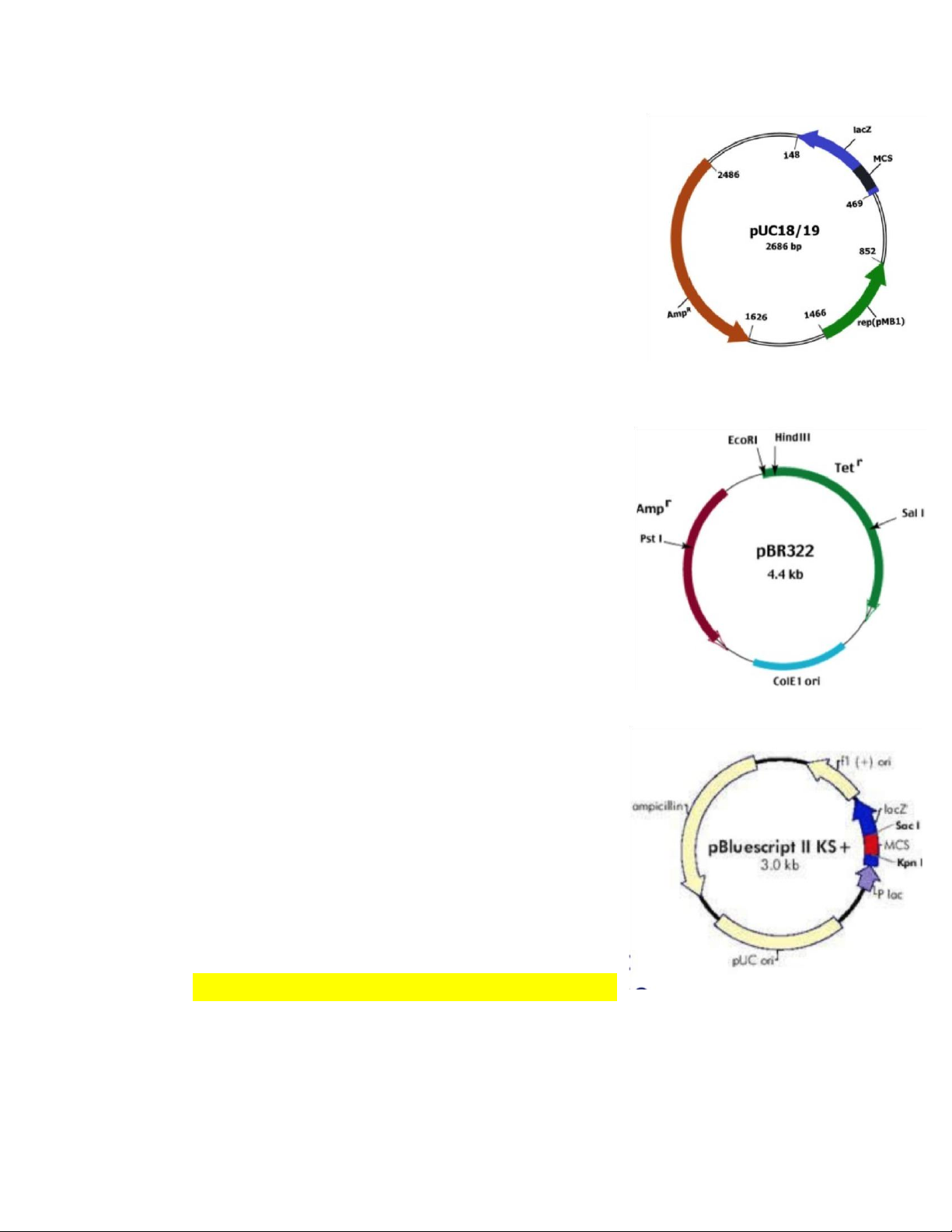
hay thẩm điện (electroporation). Một số Plasmid - pUC lOMoAR cPSD| 45469857
o Là một dãy các plasmid có mang một số
đặc tính của vector M13 và pBR, làm
thuận lợi hơn cho thao tác.
o Kích thước nhỏ (2,7 kb), có gen kháng ampicillin
o Điểm Ori của pBR322 và một phần gen lacZ của E. coli.
o MCS (tương tự M13) nằm trong đoạn
lacZ nên có thể dùng kỹ thuật bổ sung
alpha để làm yếu tố đánh dấu chèn. o
Cho số bản sao cao, và có thể được khuếch đại bằng chloramphenicol. - pBR322 o plasmid 4363 bp
o dùng tạo dòng nhanh và đơn giản các đoạn ADN
o có gen kháng tetracyline và ampicillin
o hiệu suất tạo dòng thấp hơn các vector sau này
- pGEM và pBluescript II o mang nhiều yếu tố thuận tiện cho thao tác
o kích thước khá nhỏ (2,9 kb) o cho số
bản sao cao trong tb E.coli thích hợp (SURE)
o MCS nằm giữa 2 promoter T7 và T3 cho
phép phiên mã từ trình tự được chèn vào
Các enzym biến đổi nucleotid
a. Enzym cắt hạn chế/ methylase - Cấu tạo
o Enzym cắt hạn chế (rétriction enzym – RE) là các endonuclease có
khả năng cắt DNA tại hay gần một trình tự nhận diện đặc hiệu - Chức năng lOMoAR cPSD| 45469857
o Cắt và thoái hoá các DNA ngoại lai (nd DNA phage) giúp cho bộ
gen của vi khuẩn được ổn định
- Phân loại RE: có 3 loại o Loại I và III mang một phứ hệ gồm cả 2 hoạt
tính cắt giới hạn và methyl hoá, loại này cần ATP để có năng lượng và cắt
ADN tại vị trí khá xa điểm nhận diện. Hai loại này được dùng ít trong các SHPT.
o Loại II chỉ có hoạt tính endonuclease, không cần ATP để hoạt động
và cắt ADN tại hoặc rất gần vị trí nhận diện. Loại II được sử dụng
nhiều trong thực tiễn SHPT và hiện người ta đã biết khoảng 1000
RE loại này và có vài trăm loại đã được thương mại hóa. - Danh pháp
o Dùng ba ký tự Latin (in nghiêng) trên cơ sở tên chi và loài (đôi khi
sử dụng thêm ký tự thứ tư để chỉ chủng (strain) hoặc plasmid)
o Một số La mã để chỉ thứ tự phát hiện ra enzym.
o BamHI: là RE của Bacillus amyloliquefaciens chủng H, được phát hiện đầu tiên.
Vị trí cắt của RE
- Vị trí nhận diện của đa số RE loại II thường chứa 4 – 6 nucleotid theo kiểu palindrome.
- Có 4 kiểu sắp xếp của trình tự nhận diện : o palindrome (ví dụ BamHI,
EcoRI); o palindrome bị ngắt (HinfI, XmnI);
o trình tự bị thoái biến, thường có phần còn lại của palindrome (EcoRII, HincII);
o không phải palindrome (MboII).%
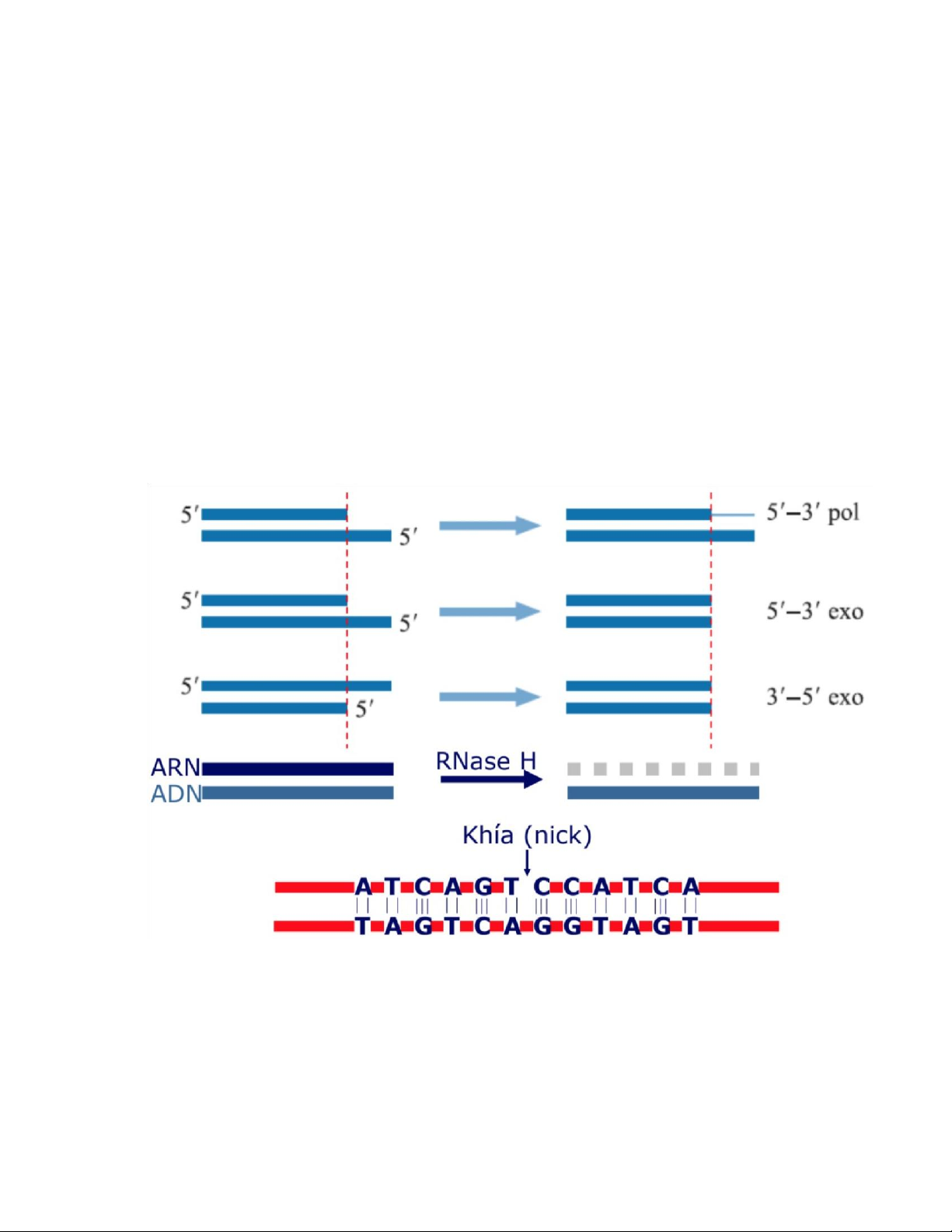
- RE chỉ cắt ADN sợi đôi và tạo ra 2 đầu tận 5’ phosphate và 3’ hydroxyl.
Đầu cắt có thể tà (tù) hoặc so le (còn gọi là đầu dính) (Hình 9.2).
- Nếu trình tự nhận diện có phần nào tính đối xứngthì điểm cắt thường
nằm giữa nó và ở vị trí giống nhau cho mỗi sợi. Ngược lại thì vị trí cắt cách khoảng so với nó. lOMoAR cPSD| 45469857
- Chỉ có hoạt tính endonuclease, không cần ATP để hoạt động và cắt ADN
tại hoặc rất gần vị trí nhận diện
- Trình tự nhận diện cắt gồm 4-8 nucleotid, thường có kiểu palindrome, VD: GAATTC - Kiểu cắt:
o Đầu tù: cắt 2 mạch tại cùng một điểm o Đầu so le/
dính: cắt mỗi mạch tại một điểm khác nhau - Ứng dụng:
o Cắt ADN trong kỹ thuật tái tổ hợp di truyền o Phân tích
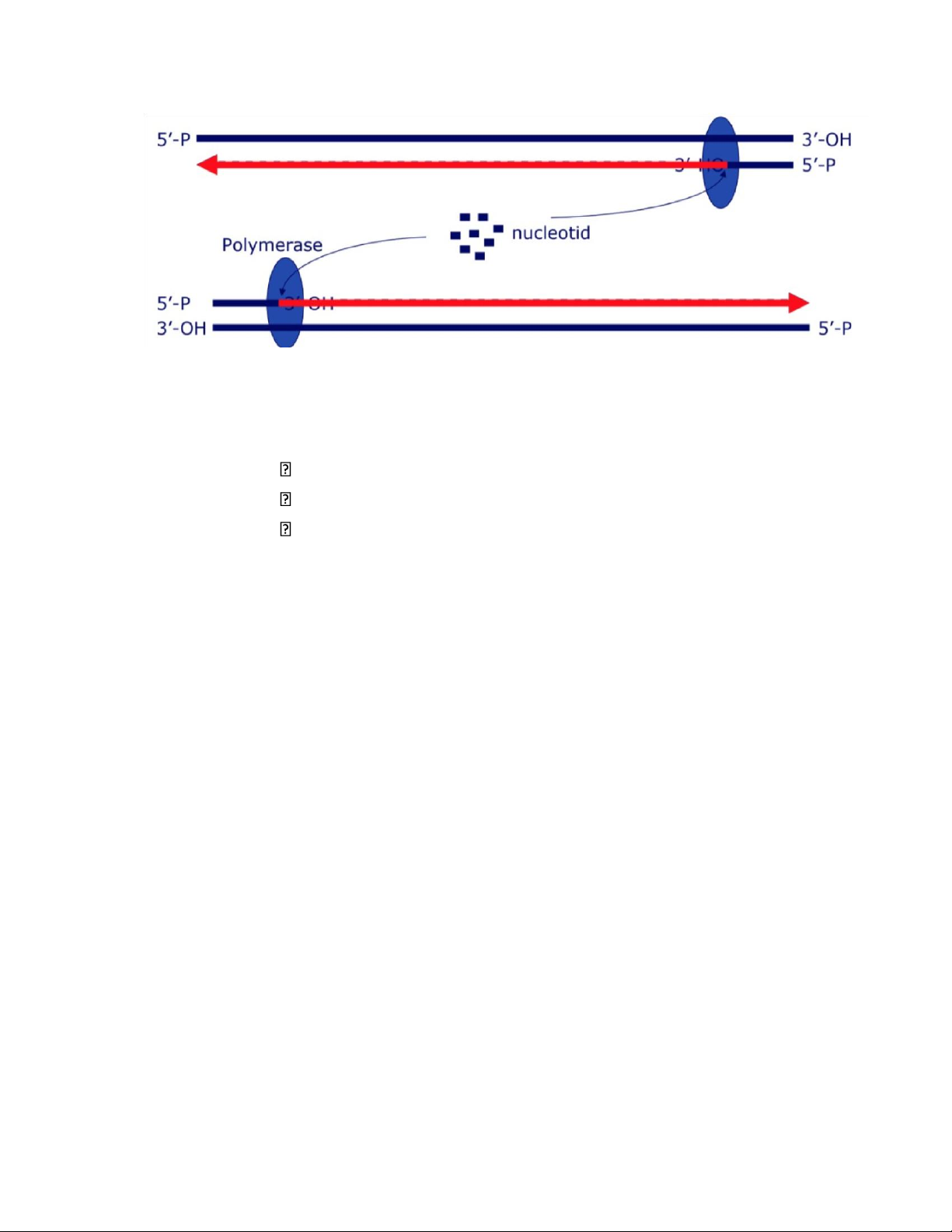
đa hình cắt giới hạn à định danh, phân biệt b. Polymerase
- Phân loại dựa trên sp và khuôn mẫu o ADN pol phụ thuộc ADN: enzym
sao chép o ARN pol phụ thuộc ADN: enzym phiên mã o ADN pol phụ
thuộc ARN: reverse transcriptase
o Polymerase không phụ thuộc khuôn mẫu: Primase
- Là enzym tổng hợp acid nucleic từ các đơn vị nucleotid
- Đặc tính chung o Hoạt tính tổng hợp 5’- 3’: Hoạt tính kéo dài. Dùng
dNTP để gắn thêm 1 deoxynucleotid-5’-monophosphate vào đầu 3’ OH
của mồi và phóng thích ppi. Tùy theo enzym, phản ứng cần có khuôn
mẫu ADN, ARN, hay không. o Hoạt tính exonuclease 3'- 5': chức năng
“sửa lỗi”. Khả năng này đối với mạch đơn mạnh hơn. Trên mạch kép nếu
có đủ dNTP, hoạt tính tổng hợp mạnh hơn, mạnh lên khi tăng nhiệt độ. lOMoAR cPSD| 45469857
o Hoạt tính exonuclease 5’- 3’: phân hủy đoạn mồi hay khuôn mẫu.
o Hoạt tính ribonuclease H: Chỉ có ở một số polymerase, nó phân
hủy đặc hiệu ARN khi có sự hiện diện của phức lai ARN/ADN.
o Dịch chuyển sợi: Một số polymerase có khả năng chuyển sợi cũ từ
vai trò khuôn mẫu thành sợi mới đang được tổng hợp. Nếu không
có hoạt tính exonuclease 5’- 3’, sự dịch chuyển sợi sẽ tạo ra sự
tổng hợp chồng chéo trên cùng một khuôn mẫu. ADN sau khi tách
ra sẽ phân hủy bởi exonuclease 3'- 5'. o Khả năng xử lý: Là giá trị
chỉ khả năng của 1 phân tử polymerase có thể tiếp tục tổng hợp
trên sợi khuôn trước khi tách ra và có thể bị thay thế bởi một polymerase khác.
o Tần suất lỗi: Khi sử dụng in vitro tần suất này thường cao hơn in
vivo và phụ thuộc vào điều kiện tiến hành. Bên cạnh đó nó cũng
tùy vào khả năng “sửa lỗi” và xử lý của polymerase.
Đặc điểm và ứng dụng của ADN polymerase I
- Là ADN pol phụ thuộc ADN
- Được phân lập từ E. coli, nhưng ngày nay đã được sản xuất từ chủng vi
khuẩn có khả năng siêu tổng hợp enzym này. lOMoAR cPSD| 45469857
- Mục đích sử dụng o Chuyển khía (nick translation): ứng dụng trong đánh dấu acid nucleic
o Tổng hợp sợi thứ 2 của cADN
- Đặc tính o Hoạt tính tổng hợp 5’- 3’: dùng đoạn mồi ADN hay ARN.Hoạt
động tốt trên khuôn mẫu ADN. Khả năng xử lý thấp và cần tối thiểu 1
mM dNTP. o Exonuclease 3'- 5': yếu hơn polymerase của T4 và T7. o Exonuclease 5’- 3’ Đoạn Klenow
- Thu được nhờ tác động của enzym thủy phân protein trên polymerase I
của E. coli hoặc nhờ siêu tổng hợp ở dòng tái tổ hợp gen qui định
polymerase đã được biến đổi. Đoạn Klenow không có hoạt tính exonuclease 5’- 3’.
- Công nghệ di truyền đã tạo được dòng sản xuất đoạn Klenow không có
hoạt tính exonuclease (exo- Klenow enzym; Stratagene). - Sử dụng:
o Làm tà đầu 3’bị hụt (đầu dính) o Loại bỏ đầu 3’ nhô ra. o Đánh
dấu đầu 3’ của ADN sợi đôi, đặc biệt đối với đầu 3’ bị hụt. o
Tổng hợp sợi thứ 2 của cADN, có ưu điểm hơn polymerase I vì
không có hoạt tính exonuclease 5’- 3’. o Đánh dấu bằng kỹ
thuật mồi ngẫu nhiên. o Giải trình tự ADN bằng phương pháp
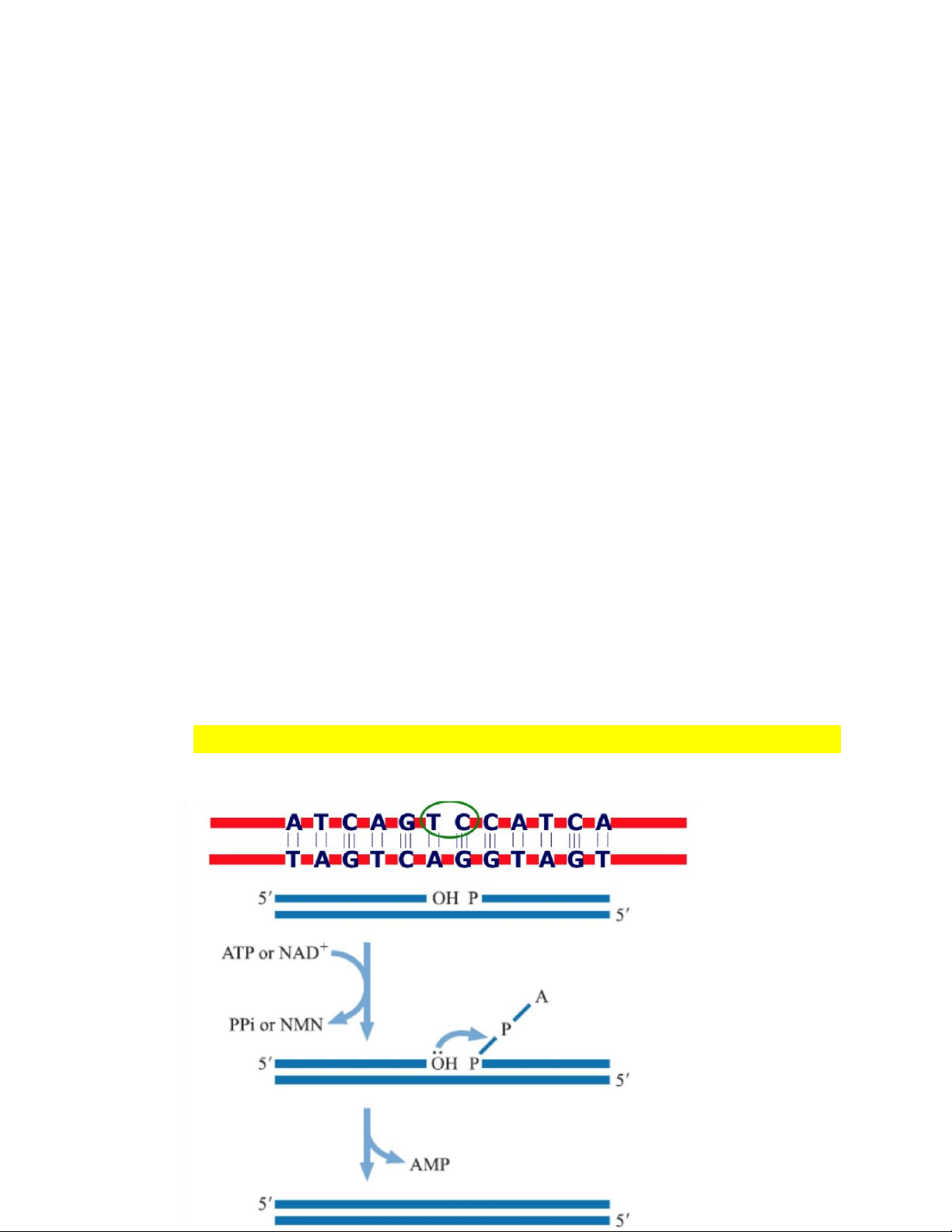
dideoxy. o Tổng hợp ADN từ ADN sợi đơn với mồi đặc hiệu. c. Ligase -
Là enzym xúc tác phản ứng tạo liên kết phosphodiester giữa 3’-OH và 5’P của hai sợi acid nucleic - ADN ligase hoạt động trên ADN sợi đôi. ARN ligase hoạt động trên ARN hoặc ADN sợi đơn - ADN ligase của E. coli cần lOMoAR cPSD| 45469857
NAD để có năng lượng và có thể dùng sửa chữa chổ bị cắt khía hay nối các đầu dính -
ADN ligase của T4 cần ATP để hoạt động và có thể dùng để sửa chổ
cắt khía, nối đầu dính và cả đầu tù -
ARN ligase của T4 có thể xúc tác nối ARN à đánh dấu đầu 3’ của ARN / sản xuất đoạn dò - Phản nối của ligase: Các kỹ thuật bản I.
Chiết tách vật liệu di truyền
- Nguyên tắc chung: 3 bước o Phá vỡ tb: vật lý, hoá học o Chiết tách
acid nucleic: phenol-chloroform o Kết tủa acid nucleic: cồn tuyệt đối
- Các trường hợp cụ thể o Plasmid: mục tiêu loại NST bằng sự khác
nhau về cấu dạng - kích thước o ADN thực khuẩn: tủa phage bằng PEG, loại bỏ capsid
o Tế bào thực vật: phá tb bằng cách nghiền o Tế bào
động vật: phá tb bằng enzym – chất tẩy o ARN: bất
hoạt Rnase, tủa= LiCl 8M, loại DNA = Dnase o Tủa
lượng ADN nhỏ: độn = tARN o Tủa = isopropanol, tert-butanol,..
- Các kỹ thuật khác o Loại carbohydrat, lipid = CTAB o Thu hồi DNA = sắc ký hấp thụ
- Yêu cầu không lẫn ADN NST
- Dựa trên một vài khác biệt về vật lý giữa ADN plasmid và ADN nhiễm
sắc thể, mà chủ yếu là về kích thước và cấu dạng (conformation). Chiết tách Plasmid
- Tách dựa trên kích thước o Phá vỡ tb trong đk được kiểm soát chặt
chẽ và êm dịu o ADN NST ko bị đứt đoạn nên có kích thước rất lớn và
thưuofng gắn với màng tb => loại bỏ = cách ly tâm
- Tách dựa trên cấu dạng – pp thuỷ giải kiềm o Tế bào bị phá vỡ với tác
nhân tẩy ion hóa như SDS, khi đó các ADN lớn như nhiễm sắc thể bị đứt đoạn.
o pH dung dịch được nâng lên 12 - 12,5 bằng NaOH làm cho các lOMoAR cPSD| 45469857
ADN thẳng bị tách thành sợi đơn, o ADN plasmid ở dạng
supercoil (siêu xoắn) nên không bị tác động. o Khi thêm acid để làm
hạ pH, ADN sợi đơn lại tái hợp, nhưng vì sợi quá dài nên kết hợp
không chính xác tạo nên bó rối dày đặc, không tan và có thể dễ dàng loại bỏ bằng ly tâm.
o Phương pháp này còn thuận lợi hơn vì trong điều kiện ly giải như
vậy, hầu hết protein và ARN cũng trở thành không tan và bị loại bỏ. Tinh chế acid nucleic
- Ly tâm phân đoạn: dung dịch acid nucleic được siêu ly tâm trong thang
tỷ trọng đường saccarose hay CsCl2 khi đó các phân tử khác nhau sẽ
tách theo tỷ trọng (tỷ lệ với kích thước phân tử). Cách này được áp dụng
khi lượng acid nucleic cần tinh chế lớn.
- Điện di gel và sắc ký lọc gel: tách acid nucleic theo kích thước phân tử ở
qui mô nhỏ (điện di) hay lớn (lọc gel). Điện di với gel polyacrylamid
(PAGE) có thể tinh chế các acid nucleic với khác biệt 1 nucleotid.
- HPLC: được áp dụng chủ yếu đối với các oligonucleotid do độ phân giải lên đến 1 nucleotid.
- Sắc ký hấp phụ: ARNm thường tận cùng bằng polyA, do đó được hấp
phụ bằng resin có gắn oligo dT hay dU.
Định tính - định lượng acid nucleic
- Quang phổ kế: OD260nm
o Nguyên tắc: dựa vào sự hấp thụ mạnh của base purin và pirimidin ở bước 260 nm
o 1 đợi vị OD260nm tương đương nồng độ:
50 μg/ml dung dịch ADN (hoặc ARN) sợi đôi
40 μg/ml dung dịch ARN (hoặc ADN) sợi đơn
30 μg/ml oligonucleotid (tới 70 base) o Kiểm tra
độ tinh khiết bằng tỷ lệ OD260/230 hoặc OD260/280 và OD320nm
o Độ tinh sạch: OD260/OD280 = 1,8 – 2.
- Điện di gel: phân tách acid nucleic = dòng điện trong gel o
Định tính theo kích thước khi so với chuẩn o Định lượng bằng
so sánh với chuẩn o Tốc độ di chuyển của ADN phụ thuộc Kích
thước ADN : tỉ lệ nghịch
Cấu dạng của ADN: tỉ lệ nghịch lOMoAR cPSD| 45469857
Nồng độ gel: tỉ lệ nghịch
Điện thế sử dụng: tỉ lệ thuận Lai acid nucleic
- Hai PT acid nucleic sợi đơn trong ĐK thích hợp có thể bắt cặp bổ sung
bằng liên kết hydro để tạo sợi đôi: quá trình này gọi là lai hóa acid nucleic (hybridization).
- PT sợi đôi thu được gọi là phân tử lai (hybrid) hay duplex.
- Quá trình ngược lại: duplex tách ra thành các sợi đơn: sự biến tính (denaturation).
- Phản ứng lai: đặc hiệu khi sự bắt cặp bổ sung hoàn toàncủa một PT,
ngược lại khi có các điểm không bắt cặp tồn tại giữa PT lai hoặc một PT
chỉ bắt cặp một phần: sự lai hóa là không đặc hiệu, không hoàn toàn.
Yếu tố ảnh hưởng lai acid nucleic
- Phản ứng lai: sự hình thành các lk hydro nên cân bằng pứ, độ đặc hiệu
và độ bền của duplex phụ thuộc vào các yếu tố sau: o Nhiệt độ
o Độ dài của các trình tự lai o Tỷ lệ trong duplex o Nồng độ ion
o Nồng độ các phân tử và thời gian pứ - Nhiệt độ chảy (Tm):
o Nhiệt độ ở đó có 50% số duplex đc hình thành
o Phụ thuộc chiều dài và tỷ lệ GC của duplex
o Nồng độ ion cx có những ảnh hưởng lớn o Tm= 4(GC) + 2(AT)
- Trong ứng dụng, phản ứng lai diễn ra giữa một phân tử đích (phân tử
cần được phát hiện) và một đoạn dò (probe).
- Đoạn dò là PT oligonucleotid đánh dấu bằng phóng xạ, huỳnh quang,
enzym hay các KT đánh dấu khác - Kỹ thuật
o Lai trên màng rắn: kỹ thuật blot, trong đó
màng nitrocellulose thường được sử dụng. lOMoAR cPSD| 45469857
Các biến thể của lai trên màng: Southern blot, Northern blot, Dot blot
o Lai tại chỗ: định vị acid nucleic đích trong mô
hay tế bào o Lai trong dung dịch: Phản ứng
được tiến hành giữa phân tử đích và đoạn dò
tự do trong DD, duplex sau đó được tách ra
khỏi dung dịch để phát hiện. Kỹ thuật này chủ
yếu áp dụng trong chẩn đoán phát hiện dựa trên acid nucleic.
- SHPT: kỹ thuật lai được ứng dụng để phát hiện sự tồn tại của acid nucleic
đích, nghiên cứu sự biểu hiện gen
- Y học: ứng dụng trong chẩn đoán phát hiện bệnh dựa trên acid nucleic và bệnh học
Southern blot, Northern blot và dot blot
- Southern blot: lai đc thực hiện với ADN sau khi đã đc điện di trong gel
rồi được chuyển và cố định trên màng
- Northern blot: lai được thực hiện với ARN sau khi đã đc điện di trong gel
rồi được chuyển và cố định trên màng
- Dot blot: lai được thực hiện với hỗn hợp acid nucleic đích được chấm trực tiếp lên màng.
Nguyên tắc kỹ thuật PCR: sao chép ADN in vitro
- Kỹ thuật PCR dựa trên đặc điểm
của quá trình sao chép gen trong tb
- Khuôn ADN sợi đơn được gắn
mồi nhờ lai hóa đặc hiệu
- Trong điều kiện có sẵn nucleotid,
ADN polymerase sẽ kéo dài mồi
theo nguyên tắc bổ sung với
khuôn ADN sợi đơn nếu đầu
3’OH của mồi còn trống tạo
thành sản phẩm sợi đôi
- Phản ứng được quay vòng nhờ
mồi thừa và sử dụng nhiệt độ
cao để tái tạo khuôn ADN sợi
đơn từ sản phẩm sợi đôi lOMoAR cPSD| 45469857
PCR – các yếu tố kỹ thuật
- Chương trình PCR o Biến tính khởi đầu: 95oC/ 5-10 phút o Chu kỳ
nhiệt: gồm 3 bước lặp lại 25-40 lần
Biến tính: 94-95oC trong 30 giây đến vài phút
Gắn mồi: 40-70oC (< Tm của mồi) trong 30 - 60 giây
Kéo dài: được thực hiện ở nhiệt độ gần với nhiệt độ tối
thích của enzym (thường là 65-74oC đối với enzym
polymerase chịu nhiệt), thời gian tùy theo độ dài khuôn
(thường 1 phút cho mỗi kb) o Kéo dài kết
thúc: 72-74oC / vài lần thời gian kéo dài
- Thành phần phản ứng: 20-100 μl
o Khuôn mẫu: 1 pg - 1 μg, nồng độ cao sẽ ức chế phản ứng.
o Mồi: 0,1 - 1 μM, nồng độ quá cao sẽ dẫn đến bắt cặp sai và làm giảm tính đặc hiệu.
o Đệm: cung cấp pH thích hợp cho phản ứng và có nồng độ Mg2+
thay đổi (cần tối ưu hóa), thường trong khoảng 0,5-5 mM. o Hỗn
hợp các dNTP: A, T, G và C, nồng độ 200-250 μM o ADN
polymerase chịu nhiệt: 0,5 - 2,5 đơn vị cho mỗi phản ứng o Nước
vừa đủ. ADN Polymerase - Taq ADN polymerase
o Ly trích từ Thermus aquaticus, hiện nay đã có dạng tái tổ hợp. o
Hoạt động tối ưu ở 75 - 80oC, hoạt tính giảm 10 lần ở 37oC.
o Hoạt tính enzym vẫn còn hơn 50% sau 2 giờ ở 95oC. Mồi trong phản ứng PCR
- Gồm mồi xuôi và mồi ngược - Là oligonucleotid lOMoAR cPSD| 45469857
- Chiều dài 18-30 (tối thiểu 15, có thể dài hơn 30) nu, có trình tự bổ sung
đặc hiệu với khuôn mẫu
- Vị trí bắt cặp trên khuôn mẫu của hai mồi sẽ quyết định vùng ADN được
khuếch đại trong PCR: Khoảng cách các vị trí bắt cặp giữa hai mồi không
quá 10 kb (tốt nhất là <3kb, một số polymerase đặc biệt có thể kéo dài đến 40kb)
- Tỷ lệ GC chiếm 40-60%. Các nucleotid AT và GC được phân bố đều, tránh
dồn vào một chỗ. Đầu 3’ của mồi nên có các nucleotid G hoặc C. Trình tự
của mồi được lựa chọn sao không tạo cấu trúc kẹp tóc và hai mồi không bắt cặp với nhau
- Nhiệt độ chảy của hai mồi phải gần nhau và trong khoảng 40-65oC,
nhưng tốt nhất là 52-58oC
- Công thức tính Tm = 2(A+T) + 4(G+C)
- Nhiệt độ bắt cặp = Tm - 5oC (cần được tối ưu)
Kiểm soát ngoại nhiễm trong PCR
- Phản ứng dễ bị ảnh hưởng bởi sự nhiễm tạp của ADN ngoại lai và chúng
có thể được khuếch đại cùng với mẫu.
- Các ADN lạ này có thể được mang sang từ các sự khuếch đại trước đó
(amplicon) hoặc từ các nguồn khác. - Kiểm soát
o qui trình thực hiện phải được tuân thủ chặt chẻ, o kiểm tra chất
lượng enzym một cách nghiêm ngặt o sử dụng bộ pipet riêng
o các thành phần phản ứng nên được phân liều trước o khu vực
chiết tách ADN, thực hiện phản ứng và phát hiện sản phẩm nên
tách biệt và thường xuyên được chiếu UV để làm giảm acid nucleic trong không khí. Các loại PCR - PCR tổ - Nested PCR
- PCR đa thành phần – Multiflex PCR - Reverse Transcriptase -PCR - Realtime PCR Realtime PCR
- Phát hiện và định hướng sản phẩm PCR = huỳnh quang theo thời gian
thực trong tube kín o Nhuộm xen giữa: lOMoAR cPSD| 45469857
Phản ứng PCR được bổ sung chất nhuộm xen giữa như SYBR Green I
Chất này nếu tự do thì không phát huỳnh quang, được chèn
vào sợi đôi ADN thì sẽ phát huỳnh quang.
Tín hiệu được đọc khi chu kỳ PCR kết thúc.
o Phân huỷ đoạn dò
1 đoạn dò đặc hiệu với trình tự sản phẩm được thiết kế: 1
đầu gắn phân tử phát huỳnh quang, 1 đầu gắn phân tử dập:
khi còn nguyên vẹn hoặc gắn trên trình tự đích thì không thể phát huỳnh quang.
Trong pha kéo dài của PCR, Taq polymerase sẽ sử dụng hoạt
tính 5’ exonuclease để phân hủy đoạn dò, phân tử phát
huỳnh quang và phân tử dập không còn ở gần nhau nên
huỳnh quang sẽ được giải phóng.
Tín hiệu được đọc khi chu kỳ PCR kết thúc.
o Lai hoá đoạn dò:
Hai đoạn dò có gắn phân tử phát huỳnh quang được thiết
kế sao cho đều gắn đặc hiệu với trình tự đích (sản phẩm
PCR) theo kiểu đuôi-đầu.
Khi cùng lai hóa trên trình tự đích huỳnh quang của phân tử
thứ nhất sẽ kích thích sự phát quang của phân tử thứ 2 tạo
ra tín hiệu, nhưng trong pha kéo dài, Taq polymerase sẽ
tách 2 đoạn dò ra thành dạng tự do nên tín hiệu sẽ mất đi.
Tín hiệu được đọc khi bắt đầu chu kỳ PCR sau bước gắn mồi. Ứng dụng PCR
- Tạo đoạn ADN mong muốn - Giải trình tự - Gây đột biến
- Chẩn đoán, phát hiện và định danh sinh vật - Xác định quan hệ di
truyền - Nhận dạng tội phạm,..
Nguyên tắc các pp đưa gen vào tế bào
- Biến nạp: dựa vào khả năng đánh bắt ADN của một số vi khuẩn
- Thẩm điện: dùng điện thế cao làm thủng màng tế bào để ADN lọt qua
- Tải nạp: sử đụng thực khuẩn để gây nhiễm tb đích lOMoAR cPSD| 45469857
- Chuyển nhiễm: dựa trên khả năng đánh bắt phức của ADN với một số
chất trong chuyển nhiễm, áp dụng chủ yếu cho tb động vật
- Đạn sinh học: gắn ADN vào hạt mang và bắn xuyên qua màng tb
Biến nạp để đưa gen vào tế bào
- Chuẩn bị tế bào khả nạp:
o Xử lý với CaCl2 (thg sd nôdng độ 50-100mM). Tại thời điểm sau xử
lý, ADN chỉ dính ở mặt ngoài tế bào chứ chưa vào trong
o Té bào khả nạp phải được bảo quản lạnh và khi sd để ở nhiệt độ thấp (4oC)
- Đưa ADN vào tế bào chất o Gây sốc nhiệt tb để việc đánh bắt ADN diễn ra
o Sốc nhiệt: mtr tb được nâng lên nhiệt độ 42oC trong thời gian
ngắn (2 phút) rồi làm lạnh nhanh
PP giải trình tự Maxam và Gilbert
- Pp hoá học: dựa vào sự thuỷ giải đặc trưng của ADN - Quy trình:
o Tạo ADN sợi đơn từ đoạn ADN cần giải trình tự o
Đánh dấu các phân tử ADN ở một đầu o Thực
hiện 4 (5) ống pứ riêng biệt:
1. G: + dimethyl sulphate => Guanine bị methyl hóa
2. AG: + formic acid => Adenin và Guanine bị methyl hóa
3. TC: + Hydrazine => biến đổi Thymin và Cytosin
4. C: + Hydrazine + 1,5 M NaCl => biến đổi Cytosin o
Xử lý 4 ống trên với piperidine 1M/90oC để cắt ADN ở liên kết
phosphat kế nucleotid bị biến đổi
o 5. A>C: NaOH 1,2N/90oC cắt ADN ở Adenin > Cytosin
- Mỗi ống được nạp vào một giếng điện di
- Kết quả điện di được đọc và biện luận trình tự PP giải trình tự Sanger
- Phương pháp enzym/dideoxy: dựa vào sự ngừng tổng hợp ADN khi gặp
phải dideoxy nucleotid (Frederick Sanger) - Qui trình:
o Thực hiện 4 phản ứng PCR riêng biệt với 1 mồi: 1.A: có dNTP + ddATP 2.T: có dNTP + ddTTP lOMoAR cPSD| 45469857 3.G: có dNTP + ddGTP
4.C: có dNTP + ddCTP o Mỗi ống sản phẩm được
nạp vào một giếng điện di
o Kết quả điện di được đọc à trình tự
Kỹ thuật tự động (Harold Swerdlow)
- Các ddNTP được đánh dấu huỳnh quang với các màu khác nhau
- Thực hiện một phản ứng PCR duy nhất với sự hiện diện của các dNTP và ddNTP
- Sử dụng điện di mao quản để tách sản phẩm
- Đọc kết quả bằng đầu dò laser




