








Preview text:
GENOMICS Bộ gen 1. Khái niệm
Bộ gen là toàn bộ bộ DNA hoàn chỉnh của sinh vật.
● Bộ gen của người là toàn bộ trình tự DNA của 1 cá thế ở tất cả cặp NST, ~ khoảng 3 tỷ nt(bp)
○ 23 cặp NST (dạng thẳng): - 22 cặp NST bình thường - 1 cặp NST giới tính
○ Có DNA trong ty thể (dạng vòng)
● DNA của vi khuẩn ~ vài triệu bp, đa phần là dạng vòng, một số là dạng thẳng.
● DNA của virus ~ vài ngàn bp
● DNA của thực vật ~ 150 tỷ bp (Vd: Paris japonica)
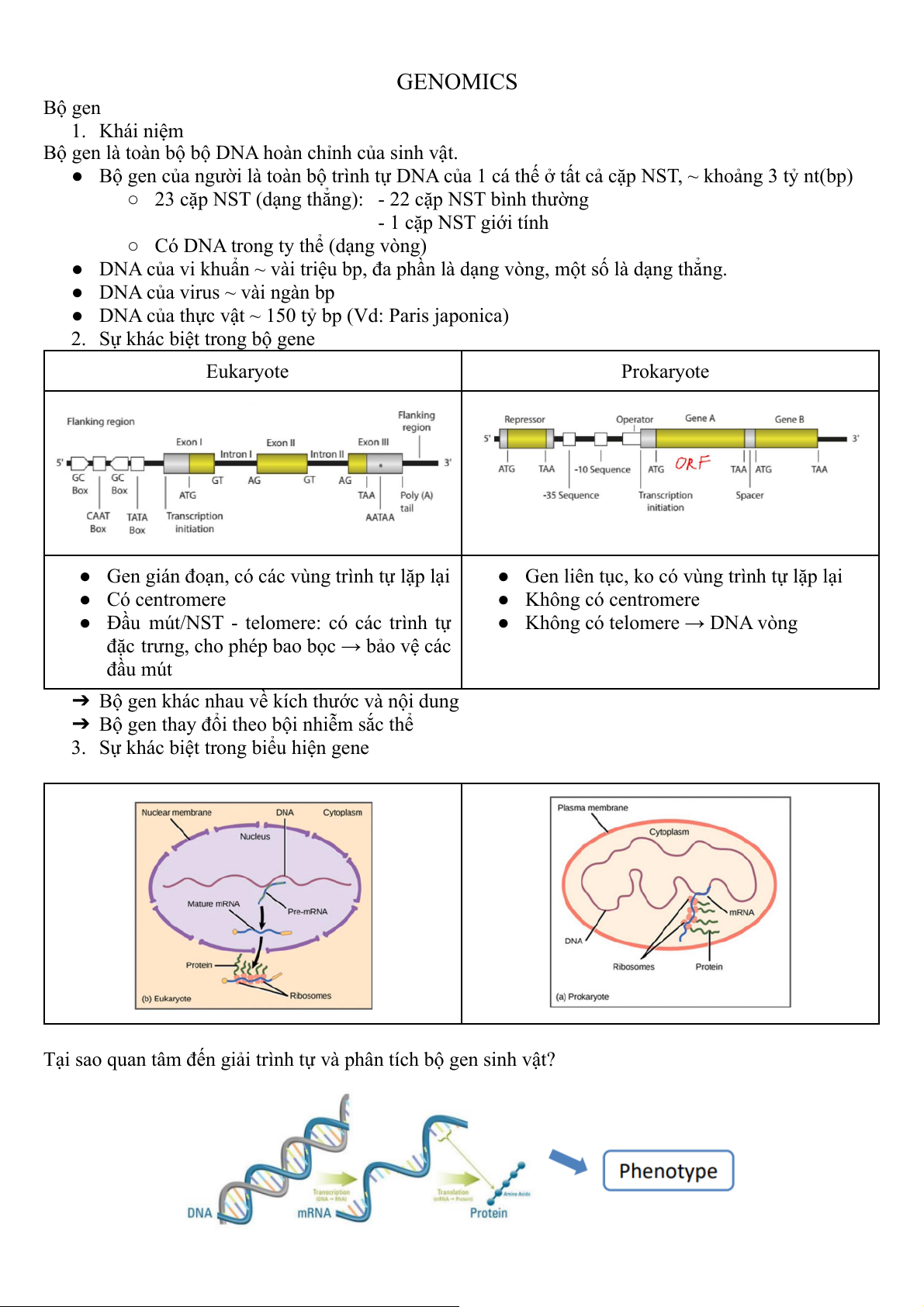
2. Sự khác biệt trong bộ gene Eukaryote Prokaryote
● Gen gián đoạn, có các vùng trình tự lặp lại
● Gen liên tục, ko có vùng trình tự lặp lại ● Có centromere ● Không có centromere
● Đầu mút/NST - telomere: có các trình tự
● Không có telomere → DNA vòng
đặc trưng, cho phép bao bọc → bảo vệ các đầu mút
➔ Bộ gen khác nhau về kích thước và nội dung
➔ Bộ gen thay đổi theo bội nhiễm sắc thể
3. Sự khác biệt trong biểu hiện gene
Tại sao quan tâm đến giải trình tự và phân tích bộ gen sinh vật?
DNA được phiên mã → mRNA → dịch mã → Protein → kiểu hình
1. Công cụ cho khoa học cơ bản
Cấu trúc/lập bản đồ gen: Trình tự bộ gen là gì? Gen nằm ở đâu Chức năng:
Tất cả DNA/gen làm việc gì? Tương tác gen ntn?
Làm sao để tế bào nhận biết DNA đang bật/tắt? 2. Công cụ y học
Nghiên cứu bộ gene → xác định, giải trình tự các gen cấu thành → áp dụng trong y học, dược phẩm, nông nghiệp,...
3. Genomics - được hình thành từ công nghệ
- Giải trình tự Sanger: 1977-1990s (tốn nhiều thời gian, chi phí)
- Giải trình tự thế hệ thứ 2 (NGS): 2007 (giải các đoạn ngắn)
- Giải trình tự thế hệ thứ 3: 2010 (giải được các đoạn dài)
➔ Giá cả của việc giải trình tự ngày càng rẻ.
CÁC CÔNG NGHỆ GIẢI TRÌNH TỰ
1. Giải trinh tự gen theo Sanger a. Nguyên tắc PCR b. Nguyên tắc Sanger
● Sử dụng các deoxy nucleotide đã bị mất nhóm hydroxyl ở đầu 3’ của phân tử đường → ddNTP
● Khi gắn ddNTP vào chuỗi → quá trình tổn hợp sẽ dừng lại
○ Nhóm 3’-OH đóng vai trò cho phép gắn thêm một nucleotide mới vào chuỗi. Nếu gắn vào
1 ddNTP không có nhóm 3’-OH → một nucleotide không được thêm vào → phản ứng
tổng hợp sẽ dừng lại. Enzyme polymerase xúc tác phản ứng gắn các dNTP vào mạch đơn
của DNA để kéo dài mạch ở vị trí 3’- OH và dừng lại nếu gắn các ddNTP vào chuỗi.
● Phản ứng gồm cả dNTP và ddNTP ⇒ hình thành vô số đoạn DNA có độ dài khác nhau do
ddNTP gắn ngẫu nhiên làm dừng phản ứng.
● Dựa vào sự sai khác về độ dài các đoạn DNA hiển thị trên bản gel sau điện di để xác định trình tự trong gene.
● Giải trình tự DNA theo Sanger ~ kỹ thuật PCR
● Thành phần của phản ứng bao gồm:
○ Enzyme DNA polymerase: xúc tác phản ứng kéo dài mạch;
○ Primer: là một đoạn DNA sợi đơn ngắn kết hợp với DNA, là trình tự khởi đầu cho polymerase; ○ Trình tự DNA
○ 4 loại deoxy nucleotide DNA (dATP, dTTP, dCTP, dGTP).
○ 4 loại dideoxy nucleotide (ddATP, ddTTP, ddCTP, ddGTP) được đánh dấu đồng vị phóng xạ.
Trong thí nghiệm của Sanger, sử dụng 4 ống nghiệm chứa 4 loại ddNTP khác nhau được đánh dấu đồng vị phóng xạ đồng
thời được thực hiện phản ứng tổng hợp.
Sau đó sản phẩm của 4 ống nghiệm được điện di hiện hình đồng vị phóng xạ trên cùng một bản gel để quan sát các band DNA.
Căn cứ vào vị trí các vạch quan sát được trên bản gel mà xác định trình tự các nucleotide theo chiều từ 5 -3 từ dưới lên trên.
Ngày nay người ta sử dụng các máy giải trình tự động để sắp xếp trình tự các band điện di. Kỹ thuật giải trình tự DNA tự
động (Dye temination sequencing) sử dụng các ddNTP có đánh dấu huỳnh quang đã giúp cho việc sắp xếp trình tự DNA trở
nên nhanh chóng và hiệu quả hơn. Mỗi loại ddNTP được gắn một màu có bước sóng phát xạ huỳnh quang khác nhau cho
phép thực hiện giải trình tự trong một phản ứng duy nhất.
Hệ thống điện di mao quản được sử dụng để đọc trình tự DNA một cách tự động. Cấu tạo của máy gồm 16 mao quản chứa
gel polyacrylamide cho phép phân tích nhiều mẫu trong một lần điện di. Hệ thống detector gồm các camera có chùm tia laser
đi qua nó để ghi nhận tín hiệu huỳnh quang một cách chính xác.
Nguyên tắc hoạt động của máy là trong suốt quá trình điện di, khi có một vạch điện di đi qua chùm tia laser thì vạch điện di
sẽ phát sáng và được camera ghi nhận và lưu lại thành một cường độ đỉnh sáng (peak) trong biểu đồ. Từ biểu đồ của các đỉnh
cường độ sáng này, máy sẽ so dòng của các đỉnh tương ứng với nhau và phân tích thành trình tự của đoạn DNA.
2. Phương pháp giải toàn bộ bộ gen (WGS) A clone-by-clone
- Cắt DNA/NST thành các mảnh nhỏ = enzyme cắt giới hạn
- Tạo bản sao rồi đưa vào NST nhân tạo của
nấm men hoặc vi khuẩn (YAC BAC)
- Khoảng 40,000 bp được chuyển vào cosmid
- Chồng các trình tự (overlap) → giải trình tự gene
Tạo ra nhiều chuỗi trình tự → gây khó khăn
Dễ hơn trong việc tổ hợp Shotgun
- Gen được cắt thành rất nhiều mảnh nhỏ → tạo dòng trong vi khuẩn
- Giải mã riêng mỗi mảnh
- Chồng các trình tự lên để sắp xếp các bản sao
- Các chương trình máy tính sẽ tổ hợp được
toàn bộ trình tự bộ gen
GIẢI TRÌNH TỰ GEN THẾ HỆ MỚI 1. Pyrosequensing 454
Kỹ thuật dựa trên nguyên lý “giải trình tự bằng tổng hợp” bao gồm: khởi động một sợi DNA đã được
giải trình tự và giải trình tự sợi bổ sung bằng phản ứng với enzyme. Đây là hệ thống có thể khuếch đại
một số lượng lớn các đoạn DNA trong giếng picotiter. Nguyên lý “giải trình tự bằng việc tổng hợp”
cũng được dựa trên việc nhận biết các pyrophosphate (PPi) được giải phóng trong quá trình gắn
nucleotide, tạo ra một tín hiệu ánh sáng, hiệu quả hơn kỹ thuật kết thúc chuỗi bằng dideoxynucleotide.
⇒ Phương thức hoạt động:
454 pyrosequensing là một phương pháp tạo ra nhiều đoạn DNA có sự tương đồng cao. Đầu tiên, DNA
được cắt thánh các đoạn đầu bằng và được gắn các oligonucleotide adaptor vào cả hai đầu. Từng đoạn
này sau đó gắn với một hạt thuỷ tinh và sẽ được khuếch đại bằng PCR trong các giọt nhũ dầu-nước, tạo
ra nhiều bản sao đoạn DNA trên mỗi hạt thuỷ tinh. Sau đó các hạt thuỷ tinh được giữ trong các giếng
picotiter trên một cơ chất dạng sợi và giải trình tự.
Quá trình giải trình tự diễn ra qua 5 bước:
- Đầu tiên, sợi khuôn, một sản phẩm PCR sợi đơn, được lai với một primer giải trình tự và Sau đó
được ủ với các enzyme DNA polymerase, ATP sulfurylase, luciferase và apyrase cũng như các
cơ chất adenosine 5’ phosphosulfate (APS) và luciferin.
- Khi dNTP đầu tiên được bắt cặp bổ sung, nó được gắn với sợi DNA khuôn nhờ DNA
polymerase và kèm theo sẽ giải phóng một PPi.
- PPi được biến đổi thành ATP nhờ enzyme ATP sulfyrelase cùng với sự tham gia của APS, sau
đó một phản ứng được xúc tác bởi luciferase sẽ biến đổi luciferin thành oxyluciferin – tạo ra ánh
sáng nhìn thấy, có độ sáng tương ứng với lượng ATP tạo thành. Ánh sáng này được nhận biết
bởi một camera và phân tích bởi một chương trình phần mềm.
- Apyrase làm suy biến các nucleotide không bắt cặp và ATP. Sau quá trình làm suy biến, phản
ứng bắt đầu lại với nucleotide khác được gắn vào. Sau một quá trình liên tục, sợi DNA bổ sung
được tổng hợp xong và việc giải trình tự nucleotide được xác định từ các đỉnh tín hiệu dựa trên
“dấu vết của program”.
2. Phương pháp Illumina (Solexa) Tóm tắt: 1. Tách mạch DNA
2. Gắn DNA sợi đơn lên giá thể
3. Thêm mồi, DNA pol, nucleotide có thể phát huỳnh quang
4. Khi tổng hợp từng nu → chụp ảnh phát huỳnh quang → ghi nhận
Chụp ảnh với độ phân giải cao → phân biệt được 10 mấy triệu điểm khác nhau
→ Nhiều trình tự 1 lúc trên cùng 1 giá thể 3. Ion torrent
Kỹ thuật xác định trình tự các nucleotide trong DNA thông qua việc phát hiện tín hiệu điện do ion H+
được giải phóng ra trong quá trình tổng hợp DNA bằng các máy đo pH siêu nhỏ gắn vào chip cảm ứng bán dẫn.
● Bước 1: Tạo thư viện DNA:
● Bước 2: PCR nhũ dịch và đưa các bead vào các giếng có chip gắn máy đo pH siêu nhỏ. Mỗi đĩa
có khoảng 1,2- 660 triệu giếng tùy theo thế hệ máy.
● Bước 3: Giải trình tự các đoạn DNA đã gắn trên mỗi bead theo nguyên lý ion semiconductor.
○ 1 loại dNTP được bơm vào các giếng trên đĩa
○ DNA polymearase xúc tác cho phản ứng kéo dài chuỗi DNA
○ Từng dNTP được thêm vào chuỗi DNA sẽ giải phóng ra PPi và H+ → số lượng H+ giải
phóng sẽ tỷ lệ với số lượng phân tử của 1 loại dNTP được gắn vào chuỗi.
○ H+ → giảm pH, tăng điện tích ở khay cảm ứng bán dẫn ⇒ chênh lệch điện thế của bộ
phận truyền cảm ứng và xuất hiện dòng điện.
○ Tín hiệu điện hóa sẽ được ghi nhận bởi phần mềm máy tính.
○ Từng loại dNTP sẽ được bơm vào trong giếng phản ứng luân phiên nối tiếp nhau.
● Bước 4: Ghép nối các đoạn DNA đã được giải trình tự bằng phần mềm tin sinh để tạo ra một
trình tự đầy đủ của genome. Ưu điểm Nhược điểm Giải trình tự
Có thể thực hiện với các đoạn DNA Tốn kém và không hiệu quả cho các dự án có Sanger
tương đối dài (khoảng 900-1200 cặp quy mô lớn hơn, như trình tự toàn bộ hệ gen base). hay metagenome. Pyro-
Độ dài đọc được là 400 bp (tương đối Khó phân tích chính xác các trình tự của 1 loại sequencing
dài) với độ chính xác càng về sau càng Nucleic acid lặp lại liên tục từ 6 nu trở lên, ví 454 tăng, dụ TTTTTT.
Công nghệ cao hơn Sanger, giải trình tự Phức tạp và nhiều công đoạn
hệ gen mà không cần tách dòng tạo thư Giá thành cao so với các phương pháp thế hệ 2
viện Thư viện DNA có thể được mã hóa khác.
và tách riêng trong suốt quá trình phân tích kết quả. Illumina
Sử dụng công nghệ bán dẫn chứ không Chiều dài đọc chỉ khoảng 100-250 bp (độ (Solexa)
phải quang học để đo tín hiệu nên thời chính xác 99% tại base thứ 250 và cao hơn ở
gian nhanh, hệ thống máy gọn nhẹ. các base phía trước.
Thời gian nhanh: 2-3 giờ để phân tích hệ Các bước thao tác phức tạp, nhiều công đoạn,
gen người Giá thành hóa chất rẻ hơn so giá thành hóa chất vẫn cao so với pp Sanger
với các kỹ thuật giải trình tự NGS khác.
Khó đếm được chính xác trình tự lặp lại liên
Thư viện DNA có thể được mã hóa và tục của 1 loại nucleotide (vd 7 nucleotide A
tách riêng trong suốt quá trình phân tích khó phân biệt với 8 nucleotide A) kết quả Ion Torrent
4 loại nucleotide được gắn huỳnh quang Giá thành hóa chất vẫn cao so với Sanger và
sequencing/Io khác nhau nên có thể phân tích được các ion torrent Chiều dài đọc chỉ khoảng 200-250 n
trình tự của 1 loại nucleotide lặp lại liên (độ chính xác 99% tại base thứ 250 và cao hơn semiconductor tục ở các base phía trước) sequencing
Thư viện DNA có thể được mã hóa và Thời gian lâu: 23 giờ để phân tích hệ gen
tách riêng trong suốt quá trình phân tích người
kết quả Cộng đồng các nhà khoa học sử Giải trình tự thế hệ 3 – Single molecular
dụng hệ thống này phổ biến hơn Torrent (SMRT) (Ion) sequencing Real Time
Phản ứng giải trình tự gen tức thời đơn phân tử được thực hiện trong một lỗ kích thước Sequencing
nano với tính chất đặc biệt: cho phép quan sát tín hiệu huỳnh quang đơn phân tử khi chất
huỳnh quang tiến sát vào đáy của lỗ nano (Zero-Mode Waveguide, ZMW). Quy trình:
Genome được phân cắt bi enzym giới hạn thành các đoạn cỡ 3000- 15000 bp.
Các đoạn DNA đượ c gắn vớ i adaptor để primer có thể bắt cặp.
DNA được ủ trong các lỗ nano đã chứa DNA polymerase cố định trong đáy lỗ.
Mỗi DNA sẽ được giải trình tự theo cơ chế tổng hợp DNA vớ i 4 loại dNTP gắn 4 loại dye
huỳnh quang khác nhau ở gốc phosphate. Tại một thờ i điểm, chỉ có dNTP nào bổ sung
với trình tự quan tâm mới đến đủ gần đáy lỗ và ngay trước khi được gắn vào chuỗi, tín
hiệu huỳnh quang mà dNTP mang được ghi nhận trong một khoảnh khắc bởi camera cực
nhạy, ngay lập tức sau đó fluorescent bị cắt và trôi nổi trong môi trường.
Toàn bộ trình tự được giải của các mảnh DNA sẽ được ghép nối và phân tích số liệu bằng phần mềm tin sinh học. Oxford
Nanopore DNA Sequencing Technology Nanopore
Giải trình tự tức thời dựa trên tín hiệu điện phân tử. Phương pháp này sử dụng dòng điện DNA
để vận chuyển một mẫu chưa biết qua một lỗ đường kính chỉ 1 nm. Sequencing
Một protein nanopore được gắn lên một màng polymer không dẫn điện. Technology
Hệ thống này luôn chứa một dung dịch điện ly – khi mà đặt vào một điện trường không
đổi, một dòng điện có thể xuất hiện trong hệ thống.
Độ lớn của cường độ dòng điện qua bề mặt lỗ nano phụ thuộc vào kích thước của lỗ nano
cũng như thành phần của DNA/RNA đang mắc tại lỗ nano đó.
Khi đủ gần với lỗ, các mẫu làm thay đổi đặc trưng về cường độ dòng điện qua bề mặt lỗ.
Tổng điện tích qua lỗ bằng tích phân mặt của cườ ng độ dòng điện chảy qua bề mặt lỗ
giữa khoảng thời gian t1 và t2.
Hai loại lỗ nano đang được quan tâm là Alpha hemolysin (αHL) từ vi khuẩn và
Mycobacterium smegmatis porin A (MspA) để ứng dụng cho giải trình tự.
Hiện nay có 3 thiết bị giải trình tự ứng dụng nguyên tắc này.
MinION flow cell có kích cỡ bỏ túi, chứa tới 512 kênh nanopore.
GridION sử dụng 5 MinION đồng thời.
PromethION chứa 48 flow cell có thể hoạt động riêng rẽ, mỗi cell có đến 3000 lỗ nano. LẮP RÁP GENE
file FASTA: gồm tên gen + trình tự Định dạng > …. > ….
file fastaq (quality) gồm tên trình tự, trình tự, chất lượng → Điểm chất lượng (QC) của một trình tự thể
hiện mục độ tin cậy của tt:
- QC = 10: Thể hiện mức độ tin cậy là 90%
- QC = 20; Thể hiện mức độ tin cậy là 99%
- QC = 30: Thể hiện mức độ tin cậy là 99,9%
- QC = 40. Thể hiện mức độ tin cậy là 99,99 % Có 2 cách:
- De Novo assembly: không có định hướng (contig: trình tự giải đc → bộ gen ban đầu)
- Map do reference: sắp xếp trên bộ gene tham chiếu
● ĐỘ PHỦ SÂU (COVERAGE)
○ Độ phủ sâu: số trình tự giải được đi qua vị trí đó
○ Ở mỗi vị trí khác nhau → độ phủ sẽ khác nhau
○ Độ 𝑝ℎủ 𝑠â𝑢 = 𝑡ổ𝑛𝑔 𝑠ố 𝑛𝑢𝑐𝑙𝑒𝑜𝑡𝑖𝑑𝑒 𝑔𝑖ả𝑖 đượ𝑐 𝑐ủ𝑎 1 𝑐𝑜𝑛𝑡𝑖𝑔
độ 𝑑à𝑖 𝑐ủ𝑎 𝑐𝑜𝑛𝑡𝑖𝑔 Ví dụ:
1. Giải được 2A và 3G → có thể là giải sâu → tra Q - độ tin cậy ⇒ Độ phủ sâu chừng 5X
(2A+3G) → nâng độ phủ sâu 50 (2A+48G)
2. Sắp gióng cột được 400,000 trình tự → 1 trình tự ~300bp, tổng độ dài contig = 2,000,000 bp
Độ 𝑝ℎủ 𝑠â𝑢 = 400,000 * 300 𝑏𝑝 = 60𝑋 2,000,000 𝑏𝑝
3. Giải trình tự → độ phủ sâu là 4X, có 2 trường hợp 4A và 3A + 1T
Để xét có sự sai xót không → nâng độ phủ sâu lên 100X
- Nếu có 99A + 1T → T là sự sai sót
- Nếu xuất hiện 75A + 25T ~ 3:1 → hoàn toàn đúng với kết quả (ko phải sai sót)
4. Độ phủ sâu của 1 vị trí → số trình tự được giải đi ngang qua đường thẳng chỉ vị trí đó
● ĐỘ PHỦ NGANG/ĐỘ PHỦ RỘNG
○ Độ phủ rộng: tỷ lệ % trình tự tham chiếu có các trình tự giải bao phủ
○ Tăng độ phủ rộng → tăng trình tự có thể giải được
Ví dụ: như trên, biết có 2 vùng không có trình tự giải được bao phủ dài 100,000 và 200,000 bp
Độ 𝑝ℎủ 𝑟ộ𝑛𝑔 = 2,000,000 − 100,000 − 200,000 = 85% 2,000,000 𝑏𝑝 TRÌNH TỰ LẶP LẠI
Vùng lặp lại (repeat regions) có chiều dài (bp) lớn (hơn tt giải đc của thế hệ t2)
⇒ Sự ra đời của Giải trình thự thế hệ thứ 3: giải được trình tự dài, trung bình giải được vài ngàn bp
(bây giờ nó giải được vài chục ngàn bp) ⇒
Ưu: chi phí thấp, giải nhanh, giải được trình tự dài
Nhược: tỷ lệ sai sót > 30%
PHÂN TÍCH CHỨC NĂNG CỦA BỘ GENE Phân tích trình tự DNA
Dựa vào các ứng dụng tin sinh học và cơ sở dữ liệu bộ gen: Sau khi cắt có 2 TH: - Cắt hoàn chỉnh
- Có các đoạn gap, đoạn contig
⇒ chú giải bộ gen: xác định các vùng chức năng của bộ gen
Trong các vùng đó → CDS (coding sequence): bao gồm vùng mở đầu (AUG) - … - bộ ba kết thúc (UAA, UAG, UGA)
Nếu ở Euka: gen gián đoạn → gen dài hơn VK vì có vùng intron
● Khi phát hiện 1 loài sinh vật mới → dùng CSDL sơ cấp vì rộng kết quả hơn → cần chú giải cấu
trúc và chức năng của bộ gen/sinh vật đó.
● Nếu không tìm ra vùng tương đồng trên CSDL → phân tích trình tự DNA (cấu trúc/chức năng)
○ Tìm các vùng trình tự bảo tồn, domain bảo tồn
○ Dựa trên XSTK để dự đoán
○ Cây dựng cây phát sinh loài
○ Học máy (machine learning); tập trung các trình tự lại, tìm mối quan hệ của chúng →
đưa cho máy tính dữ liệu học nhiều thì kq càng chính xác
● Sau khi giải mã xong, nếu vùng gen đó quan trọng → sẽ thiết kế ngược phù hợp để kiểm tra
xem chức năng của vùng gen có đúng như dự đoán không CHÚ THÍCH BỘ GEN
Gene Assembly (Lắp ráp bộ gen) Bộ gen gồm:
● Gene (coding gen): chiếm <2% bộ gen con người có thể dịch mã tạo protein ● Còn lại:
○ gen cho RNA không mã hóa (rRNA, tRNA, miRNA) ○ trình tự cấu trúc ○ trình tự điều hòa
○ gen không có chức năng
Vẫn chưa chắc là có bộ gen bao gồm bao nhiều lớp trong số này. → câu trả lời phụ thuộc vào kinh
nghiệm và sự tính toán của tin sinh học (?) QUY TRÌNH
→ Genome Assembly (lắp ráp)
→ Annotation (chú thích bộ gen): computation phase (tính toán máy tính) + synthesis phase (sinh tổng hợp)
→ manual curation (tinh chỉnh = tay) → Tìm vùng lắp ráp
→ tìm cấu trúc + chủ giải về mặt cấu trúc CDS
→ tìm đâu là vùng không được mã hóa, vùng mã hóa → chủ giải chức năng + coi kết quả THỬ THÁCH
● Giải trình tự ngắn: giải …, nếu trng đoạn gap có vùng mã hóa thì bị thiếu → kết hợp nhiều công
nghệ để giải ý hạn chế này
● Nếu là sinh vật mới sẽ gặp khó khăn (chưa có mô hình trước đây)
● Tìm hiểu chức năng của bộ gen: CSDL (cập nhật CSDL)
● Cân nhất thời gian và công sức cho chú giải tự động (tìm hiểu sơ lược) và chú giải chỉnh = tay (cụ thể hơn) FUNCTIONAL ANALYSIS OF GENOMES
Để phân tích chức năng của bộ gen: cần trình tự bộ gen, tìm vị trí mã hóa k mã hóa của gen, cơ sở dữ liệu về trình tự gen
Có 2 cách tiếp cận ( k chọn 1 trong 2 mà 2 này bổ sung cho nhau)
- tìm các vị trí mã hóa và không mã hóa, tìm các khung ORF có thể có
- tìm vùng trình tự tương đồng orf so sánh vs các trình tự đã có trên cơ sở dữ liệu để xem giống bao nhiêu %
Vd: Khung đọc mở dự đoán giống 90% trình tự trên csdlà khung đọc mở dự đoán mang chức năng của
vùng gen mà nó giống 90% →bước chú giải chức năng
Để chú giải chức năng cần: cơ sở dữ liệu, công cụ tìm kiếm khung đọc mở cũng như vùng trình tự khác
của bộ gen của tb. Việc chọn csdl để chú giải cũng quan trọng ( nếu có csdl thứ cấp thì chọn thứ cấp cho chuyên biệt)
Vi khuẩn: bộ gen liên tục, không có intron xen giữa exon→ dễ chú giải hơn euka không liên tục
Genome annotation: thuật ngữ chú giải bộ gen
Chú giải bộ gen tìm mối liên kết giữa trình tự bộ gen mình thu nhận được với chức năng sinh học của nó 2 bước chú giải
- Chú giải cấu trúc ( structural annotation): dự đoán đâu là vùng mang gen, vùng intron, exon(
đối với euka), vùng mang gen, vùng mã hóa (tRNA) của proka
- Chú giải về chức năng (functional annotation): sau khi dự đoán vùng mang gen, vùng chức năng
thì so sánh với các csdl đã biết trước đây để dự đoán chức năng dựa trên sự tương đồng về mặt trình tự.
Quá trình chú giải bộ gen: dù có bộ gen hoàn chỉnh hay chỉ có những contig thì vẫn có thể chú giải
được bộ gen ( đa phần chú giải bộ gen bằng phần mềm tin sinh)
Chú giải gồm 2 bước: (làm bằng tin sinh)
- dùng công cụ tin sinh để xác định vùng chức năng
- tổng hợp các kết quả chú giải
Có những vấn đề phần mền k nhận ra(k chính xác hoàn toàn)→ đối vs những nghiên cứu quan trọng
cần bổ sung thêm bước tinh chỉnh bằng tay, nhìn từng gen 1 để tinh chỉnh ( dù mất thời gian nhưng kết quả chính xác)
Ví dụ về quy trình chú giải
Trình tự bộ gen → tìm vùng tt lặp lại (vùng bảo tồn
do k mang chức năng) → loại những vùng đó,
những vùng còn lại đi tìm khung đọc mở và các
vùng chức năng khác nhau, vùng đc phiên mã những
k đc dịch mà (nc-RNAs)→ tìm kiếm sự tương đồng
để tìm chức năng→ công cụ genome viewer để xem kết quả chú giải
Mấy cái vàng khè là khung đọc mở hay vùng mã hóa mà tin sinh cho dự đoán, mũi tên là chiều phiên mã
Công cụ chú giải gen ngoài cho thấy sơ đồ gen và chiều mã hóa còn cho thấy tên và chi tiết về các vùng mã hóa
Lưu ý trong kết hợp dữ liệu chú giải để ra kết quả chú giải:
→ Dùng tin sinh học: throughput: hiệu suất, năng suất chủ giải nhanh nhưng kém chính xác hơn chú
giải bằng tay, nhưng chú giải bằng tay phải có kinh nghiệm và kiến thức, đánh giá từng gen 1, tốn thời gian nhiều
Thử thách trong chú giải:
Nếu bộ gen giải bằng công nghệ thứ 2, trình tự giải ngắn → kết quả chú giải chưa chính xác cho bộ
gen ( khoảng trống giữa các contig: những vùng gen k giải được)
So sánh trên csdl trình tự tương đồng để gắn chức năng: nếu chưa có sv tương đồng để chú giải thì việc
gắn chức năng khó khăn→ gọi là protein giả định ( cần làm thực nghiệm để xác định chức năng)
Để kết quả chú giải đúng thì CSDL phải lớn và luôn được cập nhật
ỨNG DỤNG & NGHIÊN CỨU Helicobacter pylori (HP)
- Vi khuẩn gram âm, có hình xoắn, sống ở môi trường vi hiếu khí, lây lan chủ yếu qua đường ăn uống.
- Cư trú trong dạ dày của khoảng 50% dân số thế giới
- Hầu hết người nhiễm HP không biểu hiện triệu chứng (80%)
- Một số trường hợp người nhiễm phát triển các bệnh lý:
✓ Viêm loét dạ dày, tá tràng (1 – 10%)
✓ Ung thư biểu mô dạ dày (UTDD) (0,1 – 3%)
✓ Lymphoma dạ dày (< 0,01%)
⇒ giải trình tự bộ gen → tìm trình tự gây bệnh Mục tiêu nghiên cứu HP:
- Hầu hết nó không có triệu chứng
- Một số rất nhỏ < bị bệnh ung thư dạ dày
Các yếu tố liên quan đến khả năng gây bệnh của HP
Sự đa dạng di truyền ở HP
→ biến động di truyền ở HP,
có loài kháng kháng sinh ở mức cao




