















Preview text:
ĐẠI HỌC BÁCH KHOA HÀ NỘI
TRƯỜNG CÔNG NGHỆ THÔNG TIN VÀ TRUYỀN THÔNG ----- ----- BÁO CÁO KỸ THUẬT
Đề tài: Dự đoán nguồn gốc tổ tiên địa lý-sinh
học (BGA) sử dụng dữ liệu DNA
Học phần: IT3190 - Nhập môn học máy và khai phá dữ liệu
Giảng viên: PGS.TS Lê Đức Hậu
TS. Nguyễn Kiêm Hiếu
Nhóm sinh viên thực hiện: STT Họ và tên MSSV 1 Nguyễn Quốc Khánh 20235118 2 Nguyễn Minh Đức 20235043 3 Trần Quí Đôn 20235039 4 Phạm Hoàng Tiến 20230071 5 Nguyễn Đình Duy 20235064
Hà Nội, tháng 1 năm 2026 LỜI CẢM ƠN
Chúng em đã nhận được rất nhiều sự quan tâm, hướng dẫn và hỗ trợ quý báu từ các thầy cô
giáo hướng dẫn, bạn bè và nhà trường trong suốt quá trình thực hiện và triển khai nghiên cứu
đề tài này. Đây là nguồn động viên to lớn giúp em hoàn thành tốt bài báo cáo với nhiều kiến
thức, kinh nghiệm và kỹ năng bổ ích.
Đặc biệt, chúng em xin gửi lời cảm ơn sâu sắc đến thầy Nguyễn Kiêm Hiếu và thầy Lê Đức
Hậu - các giáo viên hướng dẫn, những người đã định hướng nghiên cứu và góp ý tận tình và
giúp chúng em hiểu rõ hơn về các khái niệm lý thuyết cũng như những phương pháp triển
khai thực nghiệm dự án, cũng như giúp đỡ chúng em trong quá trình hoàn thiện các phần của
dự án. Những chỉ dẫn tận tâm của các thầy là nền tảng quan trọng giúp nhóm hoàn thiện đề tài
một cách khoa học và hiệu quả hơn.
Mặc dù đã nỗ lực hoàn thiện bài báo cáo với tinh thần trách nhiệm cao nhất, nhưng chắc
chắn chúng em không thể tránh khỏi những thiếu sót. Vì vậy, nhóm rất mong nhận được
những ý kiến đóng góp quý báu từ quý thầy cô để có thể rút kinh nghiệm và hoàn thiện hơn
trong các đồ án, nghiên cứu sau này. Mục lục
LỜI CẢM ƠN............................................................................................................................2
Phần 1. Giới thiệu bài toán.......................................................................................................4
1.1. Bối cảnh và định nghĩa...........................................................................................4
1.2. Cơ sở khoa học và kỹ thuật................................................................................... 4
1.3. Tính cấp thiết và thách thức...................................................................................4
1.4. Mục tiêu của đề tài................................................................................................. 4
Phần 2. Dữ liệu bài toán........................................................................................................... 5
2.1. Nguồn dữ liệu.................................................................................................................5
2.2 Mô tả dữ liệu................................................................................................................... 5
2.3. Chia tập Train / Validation / Test................................................................................... 6
Phần 3. Các phương pháp thử nghiệm....................................................................................7
3.1. Tổng quan các phương pháp.......................................................................................... 7
3.2. Mô tả chi tiết.................................................................................................................. 7
3.2.1. XGBoost (Baseline).............................................................................................. 7
3.2.2. Generative Bayesian Model.................................................................................. 7
Phần 4. Hàm mục tiêu và hàm mất mát..................................................................................9
4.1. Mục tiêu tổng quát......................................................................................................... 9
4.2. XGBoost Multiclass.......................................................................................................9
4.3. Generative Bayesian Model......................................................................................... 10
Phần 5. Độ đo đánh giá........................................................................................................... 11
5.1. Accuracy...................................................................................................................... 11
5.2. Classification Report (Precision, Recall, F1-score cho từng lớp)................................11
5.3. Confusion Matrix......................................................................................................... 11
5.4. Macro-F1 (dùng trong tuning)..................................................................................... 11
5.5. Uncertainty / Max Posterior (Generative Model)........................................................ 11
Phần 6. Thiết lập thí nghiệm.................................................................................................. 13
6.1. Môi trường thực nghiệm.............................................................................................. 13
6.2. Siêu tham số (Hyperparameters)..................................................................................13
6.2.1. XGBoost (baseline train scripts)......................................................................... 13
6.2.2. XGBoost (hyperparameter tuning cho Đông Á)................................................. 14
6.2.3. Generative Bayesian Model................................................................................ 14
Phần 7. Kết quả thực nghiệm.................................................................................................16
7.1. Kết quả thực nghiệm cho bài toán phân loại cấp Châu lục....................................................... 16
7.2. Kết quả thực nghiệm cho bài toán phân loại cấp Đông Á.........................................................18
Phần 8. Đường học.................................................................................................................. 21
8.1. Đường học cho bài toán phân loại cấp Châu lục.......................................................................21
8.2. Đường học cho bài toán phân loại cấp Đông Á........................................................................ 23
Tài liệu tham khảo.................................................................................................................25
Phụ lục....................................................................................................................................................26 Phần 1.
Giới thiệu bài toán
1.1. Bối cảnh và định nghĩa
Bài toán phân tích kiểu hình DNA pháp y (Forensic DNA Phenotyping - FDP) nhằm dự đoán
nguồn gốc địa lý sinh học (Biogeographical Ancestry - BGA) là một nhánh quan trọng trong
khoa học hình sự hiện đại. Khác với phương pháp định danh truyền thống dựa trên việc đối
khớp trực tiếp các đoạn lặp ngắn (STR) trong cơ sở dữ liệu tội phạm, BGA tập trung vào việc
khai thác các đặc điểm di truyền để xác định nguồn gốc tổ tiên của một cá nhân.
Thông tin này cung cấp "mô tả sinh học" giúp thu hẹp phạm vi điều tra trong các trường hợp
không tìm thấy kết quả khớp DNA trực tiếp tại hiện trường hoặc khi nghi phạm không nằm
trong bất kỳ cơ sở dữ liệu nào hiện có.
1.2. Cơ sở khoa học và kỹ thuật
Cơ sở cốt lõi của bài toán dựa trên các chỉ thị thông tin về nguồn gốc (Ancestry Informative
Markers - AIMs). Trong đó, đa hình đơn nucleotide (SNPs) được ưu tiên sử dụng nhờ:
● Tính ổn định cao: SNPs có tốc độ đột biến thấp, giúp lưu giữ thông tin tổ tiên qua hàng ngàn năm.
● Tần suất khác biệt: Các AISNP (Ancestry Informative SNPs) có tần suất allele khác
biệt rõ rệt giữa các quần thể người ở các châu lục và tiểu vùng địa lý khác nhau do tác
động của chọn lọc tự nhiên và trôi dạt di truyền (genetic drift).
● Dữ liệu quy mô lớn: Sự sẵn có của các dự án như 1000 Genomes Project cung cấp nền
tảng dữ liệu genotype chuẩn để huấn luyện các mô hình học máy.
1.3. Tính cấp thiết và thách thức
Việc phân loại nguồn gốc tổ tiên đối mặt với hai thách thức lớn:
1. Độ nhiễu dữ liệu: Sự di cư và giao thoa giữa các quần thể khiến ranh giới di truyền trở nên mờ nhạt.
2. Độ chi tiết (Granularity): Phân loại ở cấp độ châu lục (ví dụ: Đông Á vs Châu Âu)
tương đối khả thi, nhưng phân loại sâu hơn vào các quần thể con (ví dụ: người Kinh vs
người Hán) đòi hỏi các đặc trưng tinh vi hơn và thuật toán tối ưu hơn.
1.4. Mục tiêu của đề tài
Dự án này tập trung xây dựng và đánh giá hệ thống dự đoán BGA thông qua hai tầng phân loại chính:
● Tầng 1 (Continental Level): Phân loại 5 nhóm tổ tiên chính (Châu Phi, Châu Mỹ,
Đông Á, Châu Âu, Nam Á).
● Tầng 2 (Sub-population Level): Phân loại chi tiết nội bộ khu vực Đông Á (EAS) bao
gồm 5 quần thể con (CDX, CHB, CHS, JPT, KHV).
Mục tiêu cụ thể là so sánh hiệu suất giữa mô hình Boosting mạnh mẽ (XGBoost) đóng vai trò
là baseline và mô hình xác suất sinh (Generative Bayesian Model), từ đó đánh giá khả năng
ứng dụng của các thuật toán này trong bài toán pháp y thực tế. 4 Phần 2. Dữ liệu bài toán
2.1. Nguồn dữ liệu
Danh sách AISNP (Ancestry Informative Single Nucleotide Polymorphisms) được trích xuất
từ Phụ lục Supplementary Material 1 của bài báo Systematic analyses of AISNPs screening
and classification algorithms based on genome-wide data for forensic biogeographic ancestry
inference đăng trên tạp chí Forensic Science International: Genetics (2024). Phụ lục này cung
cấp tập hợp các SNP đã được chọn lọc và đánh giá có khả năng phân biệt nguồn gốc tổ tiên
địa lý – sinh học ở nhiều cấp độ khác nhau.
Dữ liệu genotype được lấy từ 1000 Genomes Project Phase 3, bao gồm 2.504 cá thể người với
bộ gen tham chiếu hg19. Dữ liệu được tải trực tiếp từ các mirror chính thức của dự án (định
dạng VCF), là nguồn dữ liệu chuẩn thường được sử dụng trong các nghiên cứu di truyền quần
thể và suy luận tổ tiên sinh học. Việc truy xuất dữ liệu được thực hiện thông qua các script
bash, sử dụng công cụ bcftools để query chính xác các rsID thuộc danh sách AISNP đã xác định.
2.2 Mô tả dữ liệu
Dataset được thiết kế theo hai tầng phân loại (hierarchical classification), phản ánh cách tiếp
cận phổ biến trong bài toán suy luận nguồn gốc tổ tiên sinh học: từ cấp độ châu lục đến cấp độ quần thể khu vực.
Về số lượng mẫu, ở tầng châu lục (continental level), dataset bao gồm toàn bộ 2.504 mẫu từ
1000 Genomes Project, được phân bố vào 5 nhóm tổ tiên chính (super_pop): Châu Phi (AFR),
Châu Mỹ (AMR), Đông Á (EAS), Châu Âu (EUR) và Nam Á (SAS). Ở tầng Đông Á (East
Asian level), tập dữ liệu được giới hạn trong 504 mẫu thuộc nhóm super_pop = EAS, bao
gồm 5 quần thể con (pop): CDX, CHB, CHS, JPT và KHV.
Về đặc trưng (features), dataset sử dụng tổng cộng 58 SNP, trong đó:
● 24 SNP được sử dụng cho bài toán phân loại tổ tiên ở cấp châu lục.
● 34 SNP được sử dụng riêng cho bài toán phân loại nội bộ Đông Á.
Mỗi SNP ban đầu được biểu diễn bằng hai cột allele (ví dụ: rsXXXX_1 và rsXXXX_2),
tương ứng với hai bản sao nhiễm sắc thể của mỗi cá thể. Dữ liệu genotype gốc ở dạng ký tự
nucleotide (A, T, G, C) được chuyển đổi sang dạng số thông qua bước encoding theo số lượng allele minor:
● 0: đồng hợp allele major ● 1: dị hợp
● 2: đồng hợp allele minor
Cách biểu diễn này cho phép các mô hình học máy xử lý dữ liệu genotype như các đặc trưng
số, đồng thời vẫn giữ được ý nghĩa sinh học cơ bản của SNP. 5
Về kiểu dữ liệu, các trường metadata như sample, pop và super_pop thuộc dạng categorical,
đóng vai trò định danh mẫu và nhãn phân loại. Dữ liệu genotype sau encode là numeric, phù
hợp cho các thuật toán học có giám sát.
Về nhãn (labels), ở tầng 1 (châu lục), nhãn mục tiêu là super_pop với 5 lớp chính. Ở tầng 2
(Đông Á), nhãn mục tiêu là pop với 5 quần thể con. Cấu trúc hai tầng này phản ánh rõ tính
phân cấp của bài toán BGA và cho phép đánh giá khả năng phân biệt tổ tiên ở các mức độ chi tiết khác nhau.
2.3. Chia tập Train / Validation / Test
Chiến lược chia dữ liệu được thiết kế nhằm đảm bảo tính công bằng giữa các lớp và tính ổn
định của đánh giá mô hình.
Đối với cả hai tầng phân loại, dữ liệu được chia thành tập Train và Test theo tỷ lệ 80/20. Việc
chia được thực hiện theo phương pháp stratified, dựa trên nhãn mục tiêu, nhằm bảo toàn phân
bố lớp gốc của 1000 Genomes trong cả hai tập.
Trong các thí nghiệm huấn luyện tiêu chuẩn , 80% dữ liệu được dùng để huấn luyện mô hình,
trong khi 20% còn lại được giữ nguyên làm tập Test để báo cáo các chỉ số đánh giá cuối cùng như Accuracy và macro-F1.
Đối với các thí nghiệm điều chỉnh siêu tham số , nhóm áp dụng Stratified K-Fold
Cross-Validation với số fold k = 5, có shuffle và random_state = 42. Quá trình này chỉ được
thực hiện trên phần Train (80%), trong đó mỗi vòng CV ngầm chia dữ liệu thành khoảng 64%
Train và 16% Validation theo cách stratified. Chỉ số macro-F1 được tổng hợp qua các fold để
lựa chọn bộ siêu tham số tối ưu. Sau khi hoàn tất quá trình tuning, mô hình cuối cùng được
huấn luyện lại trên toàn bộ 80% tập Train trước khi đánh giá trên tập Test độc lập.
Do dữ liệu 1000 Genomes mang tính cross-sectional và không gắn với yếu tố thời gian, việc
chia tập được thực hiện hoàn toàn ngẫu nhiên với seed cố định (42), đảm bảo khả năng tái lập
kết quả giữa các lần chạy thí nghiệm. 6 Phần 3.
Các phương pháp thử nghiệm
3.1. Tổng quan các phương pháp
Dự án của nhóm sử dụng 2 mô hình: XGBoost và Generative Bayesian Model. Trong đó,
XGBoost được chọn làm mô hình Baseline.
3.2. Mô tả chi tiết
3.2.1. XGBoost (Baseline)
XGBoost là một thuật toán học máy dựa trên cây quyết định (Decision Tree) kết hợp kỹ thuật
Boosting, cực kỳ phổ biến trong các cuộc thi dữ liệu nhờ hiệu suất cao và khả năng xử lý dữ liệu bảng tabular data.
Nhóm lựa chọn XGBoost làm mô hình Baseline của bài toán, vì:
● Dữ liệu SNP thường có nhiều biến, nhưng không phải locus nào cũng có ý nghĩa cho
mọi quần thể. XGBoost xử lý rất tốt các đặc trưng thưa và không cần chuẩn hóa dữ
liệu đầu vào quá phức tạp.
● Trong pháp y, việc biết SNP nào đóng vai trò quan trọng nhất để phân biệt người
Đông Á với người Châu Âu là cực kỳ quan trọng để tối ưu hóa bộ kit xét nghiệm. Vì
vậy, khả năng xếp hạng đặc trưng (Feature Importance) của XGBoost có thể được dùng tới.
● Với lượng dữ liệu lớn từ các dự án như 1000 Genomes, XGBoost huấn luyện rất
nhanh nhờ khả năng tính toán song song.
XGBoost có các ưu điểm sau:
● Độ chính xác cao: Thường vượt trội hơn các thuật toán truyền thống như SVM hay
Random Forest trong các bài toán phân loại đa lớp.
● Kiểm soát Overfitting: Có các tham số điều tiết (regularization) giúp mô hình không bị
"học vẹt" trên dữ liệu huấn luyện nhỏ của một số quần thể hiếm.
● Xử lý phi tuyến: Có thể bắt được các tương tác phức tạp giữa các gene (epistasis) mà
các mô hình tuyến tính bỏ qua.
Bên cạnh đó, mô hình cũng có một số nhược điểm:
● Hộp đen (Black box): Mặc dù có feature importance, nhưng rất khó để giải thích "tại
sao" một tổ hợp gene cụ thể lại dẫn đến kết luận về một quần thể Đông Á chi tiết
(Chẳng hạn như phân biệt người Kinh và người Hán).
● Nhạy cảm với mất cân bằng dữ liệu: Ở tầng 2 (phân loại chi tiết), nếu số lượng mẫu
của một quần thể quá ít, XGBoost dễ bị thiên kiến (bias) về phía quần thể có mẫu lớn hơn.
3.2.2. Generative Bayesian Model
Khác với các mô hình phân biệt (Discriminative) như XGBoost, mô hình Bayes sinh tập trung
vào việc mô hình hóa phân phối xác suất của dữ liệu cho từng nhóm quần thể. Một ví dụ điển
hình trong di truyền học là thuật toán STRUCTURE hoặc các biến thể của Naive Bayes. 7
Nhóm chọn Generative Bayesian Model làm mô hình nâng cao của bài toán, vì:
● Mô hình Bayes rất phù hợp với quy luật di truyền Mendel. Nó giả định rằng mỗi cá
thể là một hỗn hợp xác suất từ các "quần thể nguồn" (ancestral populations).
● Trong thực tế, ít khi một người là "100% thuần chủng". Mô hình Bayes cho phép tính
toán tỷ lệ % tổ tiên (ví dụ: 70% Đông Á, 30% Đông Nam Á), điều này thực tế hơn so
với việc chỉ dán một nhãn duy nhất.
Mô hình Bayes có các ưu điểm sau:
● Tính diễn giải cao (Interpretability): Kết quả đầu ra là xác suất hậu nghiệm (Posterior
Probability). Các nhà pháp y có thể đưa ra kết luận kèm theo mức độ tin cậy cụ thể (ví
dụ: "Khả năng 95% mẫu thuộc về quần thể A").
● Hoạt động tốt với dữ liệu nhỏ: Nếu ta có kiến thức nền (Prior knowledge) về tần suất
allele trong các quần thể, mô hình Bayes cần ít dữ liệu huấn luyện hơn để đạt được kết
quả ổn định so với XGBoost.
● Xử lý dữ liệu thiếu: Trong pháp y, mẫu DNA có thể bị thoái hóa dẫn đến mất một số
locus. Mô hình Bayes xử lý việc thiếu dữ liệu này một cách tự nhiên thông qua xác suất điều kiện.
Bên cạnh đó mô hình cũng có một số nhược điểm:
● Giả định độc lập: Các mô hình Bayes đơn giản (như Naive Bayes) giả định các SNP
độc lập với nhau (loại bỏ liên kết không cân bằng - Linkage Disequilibrium), điều này
có thể làm giảm độ chính xác nếu không được xử lý kỹ.
● Chi phí tính toán: Các mô hình Bayes phức tạp (sử dụng MCMC - Markov Chain
Monte Carlo) tốn rất nhiều thời gian và tài nguyên để hội tụ, đặc biệt là ở tầng phân
loại chi tiết với hàng nghìn SNP. 8 Phần 4.
Hàm mục tiêu và hàm mất mát
4.1. Mục tiêu tổng quát
Bài toán yêu cầu phân loại nguồn gốc địa lý sinh học theo hai tầng (châu lục và Đông Á). Do
đó mục tiêu tối ưu hóa chung là giảm thiểu sai số phân loại (classification error) hoặc tương
đương là tối đa hóa xác suất đúng của nhãn thật. Tất cả mô hình đều học từ các mẫu đã gán
nhãn (`super_pop`, `pop`) nên đây là supervised multiclass classification.
4.2. XGBoost Multiclass
● Hàm mục tiêu: Trong bài toán phân loại đa lớp, mô hình XGBoost được cấu hình với
tham số objective="multi:softprob" trong lớp xgboost.XGBClassifier. Hàm mục tiêu
này yêu cầu mô hình học cách dự đoán phân phối xác suất trên toàn bộ các lớp, thay vì
chỉ trả về một nhãn phân loại duy nhất. Cụ thể, với mỗi mẫu đầu vào, mô hình xuất ra
một vector xác suất, trong đó mỗi phần tử biểu thị xác suất mẫu đó thuộc về một lớp
nhất định. Tổng xác suất trên tất cả các lớp bằng 1. Việc tối ưu hàm mục tiêu
multi:softprob tương đương với việc tối đa hóa log-likelihood của bài toán phân loại
đa lớp, hay nói cách khác là làm cho phân phối xác suất dự đoán của mô hình tiệm cận
nhất với phân phối nhãn thật được biểu diễn dưới dạng one-hot.
● Hàm mất mát: Hàm mất mát được sử dụng trong quá trình huấn luyện là multiclass
logarithmic loss (mlogloss), tương ứng với hàm cross-entropy đa lớp. Với mỗi mẫu
huấn luyện, loss được tính dựa trên xác suất mà mô hình gán cho lớp đúng của mẫu
đó, cụ thể là giá trị âm của logarit tự nhiên của xác suất này. Nếu mô hình dự đoán xác
suất cao cho lớp đúng, giá trị loss sẽ nhỏ; ngược lại, nếu mô hình gán xác suất thấp
cho lớp đúng, loss sẽ lớn. Tổng giá trị mất mát của mô hình được tính bằng trung bình
(hoặc tổng) loss trên toàn bộ tập dữ liệu. Trong quá trình huấn luyện, XGBoost sử
dụng gradient của hàm log-loss để cập nhật các cây quyết định, nhằm giảm dần giá trị
mất mát và từ đó gián tiếp cải thiện độ chính xác phân loại của mô hình.
● Bối cảnh áp dụng: Trong nghiên cứu này, mô hình XGBoost được áp dụng theo kiến
trúc phân loại hai tầng nhằm phản ánh cấu trúc phân cấp của bài toán dự đoán nguồn
gốc tổ tiên sinh–địa lý.
○ Ở tầng thứ nhất, mô hình được huấn luyện với nhãn super_pop, tương ứng
với 5 nhóm lục địa chính. Mục tiêu của tầng này là học ranh giới phân tách ở
mức độ khái quát giữa các quần thể lớn. Quá trình huấn luyện được theo dõi
thông qua chỉ số log-loss, đảm bảo mô hình hội tụ tốt và không bị overfitting.
Sau khi xác định cấu hình phù hợp và đạt giá trị log-loss thấp, mô hình được
huấn luyện lại trên toàn bộ tập dữ liệu huấn luyện để xây dựng phiên bản cuối
cùng phục vụ triển khai thực tế.
○ Ở tầng thứ hai, mô hình tập trung vào phân loại chi tiết hơn trong phạm vi các
quần thể Đông Á, với nhãn pop. Tại tầng này, quá trình tinh chỉnh siêu tham
số được thực hiện bằng phương pháp cross-validation, trong đó macro-F1
score được sử dụng làm tiêu chí đánh giá và lựa chọn cấu hình tối ưu. Việc sử
dụng macro-F1 nhằm đảm bảo hiệu năng cân bằng giữa các lớp, đặc biệt trong
bối cảnh phân bố dữ liệu không đồng đều. Tuy nhiên, cần lưu ý rằng bản chất
quá trình huấn luyện của XGBoost vẫn tối ưu hàm log-loss đa lớp, còn
macro-F1 chỉ đóng vai trò là chỉ số đánh giá bên ngoài để lựa chọn mô hình có
khả năng phân biệt các lớp tốt và ổn định hơn. 9
4.3. Generative Bayesian Model
Hàm mục tiêu: mô hình sinh ước lượng tần số allele pk,j cho từng population k và SNP j bằng
cách tối đa hóa log-likelihood của dữ liệu dưới phân bố Binomial với prior Beta(`alpha`,
`alpha`). Trong cài đặt này, việc fit thực chất là tính ước lượng MAP theo công thức:
và prior của lớp là tần suất mẫu (hoặc user-defined). Không có vòng lặp gradient.
Hàm mất mát / tiêu chí: không có “loss” tường minh như trong mô hình phân biệt; thay vào
đó, mô hình tối đa hóa log-likelihood khi ước lượng tham số và tại suy luận sử dụng negative
log posterior làm thước đo chất lượng dự đoán. Khi cần đánh giá bất định (uncertainty), ta
nhìn vào `max_probs` từ posterior: mẫu có `max_prob < threshold` bị gán `UNKNOWN`
(tương đương kiểm soát rủi ro bằng ngưỡng x ác suất chứ không tối ưu thêm loss mới). 10 Phần 5. Độ đo đánh giá 5.1. Accuracy
- Đây là độ đo chính xác của mô hình, được tính theo công thức sau:
Accuracy = số mẫu dự đoán đúng / tổng số mẫu
Với bài toán phân loại đa lớp, đây là tỷ lệ mẫu mà mô hình gán đúng nhãn `super_pop` (tầng 1) hoặc `pop` (tầng 2).
Lý do chọn: mục tiêu cuối cùng của pipeline là xác định đúng nguồn gốc địa lý; accuracy trực
quan, dễ so sánh với các nghiên cứu forensic khác. Trong các script huấn luyện
(`scripts/train_continental_xgb.py`, `scripts/train_eastasian_xgb.py`) accuracy được in ra
ngay sau khi dự đoán (`src/models.py:22-34`).
5.2. Classification Report (Precision, Recall, F1-score cho từng lớp)
Thư viện sklearn.metric cung cấp Precision, Recall, F1-score theo từng lớp. F1 ở đây là
harmonic mean giữa precision và recall trong từng population.
Lý do chọn: các lớp (đặc biệt trong tầng Đông Á) có số mẫu không đồng đều; xem
Precision/Recall/F1 giúp phát hiện lớp nào bị bias hoặc khó phân lớp. Đây cũng là cách giám
sát chất lượng khi triển khai cho pháp y (cần biết xác suất nhầm lẫn theo lớp). 5.3. Confusion Matrix
Ma trận nhầm lẫn (Confusion Matrix) hiển thị số lượng dự đoán lớp i thành lớp j. Mỗi hàng là
nhãn thật, mỗi cột là dự đoán.
Lý do chọn: Ma trận giúp dễ dàng nhìn ra các cặp lớp hay nhầm (ví dụ EUR vs AMR, CHB
vs CHS). Điều này quan trọng để đánh giá ứng dụng forensic: nếu một cặp lớp hay nhầm, cần
trình bày rõ trong báo cáo khoa học hoặc điều chỉnh mô hình.
5.4. Macro-F1 (dùng trong tuning)
Macro-F1 được tính bằng giá trị trung bình F1-score của tất cả lớp, không trọng số theo kích thước lớp.
Trong `scripts/tune_eastasian_xgb.py`, macro-F1 được dùng làm scoring của `RandomizedSearchCV`.
Lý do chọn: East Asian subpopulations phân bố tương đối cân bằng nhưng vẫn cần đảm bảo
mỗi lớp quan trọng như nhau. Macro-F1 tránh việc mô hình “ăn gian” bằng cách tối ưu lớp có
nhiều mẫu nhất; nó phù hợp cho bài toán forensic nơi mỗi population quan trọng ngang nhau.
5.5. Uncertainty / Max Posterior (Generative Model)
Khi dùng GenerativeBGAModel, mỗi mẫu có vector posterior probability. Ta dùng
`max_prob` (xác suất lớn nhất) làm độ đo độ tin cậy; nếu thấp hơn threshold thì gán nhãn `UNKNOWN`.
Lý do chọn: Trong ứng dụng pháp y, việc báo cáo “không chắc chắn” sẽ tốt hơn là trả về nhãn
sai. Thay vì ép mô hình chọn một lớp, metric này cho phép định lượng mức tin cậy và đưa vào báo cáo điều tra.
Tóm lại, Bộ độ đo kết hợp Accuracy + classification report + confusion matrix giúp đánh giá
toàn diện các mô hình discriminative. Macro-F1 đảm bảo cân bằng lớp trong quá trình tune. 11
Với mô hình generative, max posterior/uncertainty cung cấp góc nhìn bổ sung về độ tin cậy của dự đoán. 12 Phần 6.
Thiết lập thí nghiệm
6.1. Môi trường thực nghiệm
Ngôn ngữ lập trình: Python 3.12.11 (CPython, build từ conda-forge). Thư viện chính:
● `numpy>=1.24`, `pandas>=2.0` cho xử lý dữ liệu genotype.
● `scikit-learn>=1.3,<1.6` dùng cho `train_test_split`, `LabelEncoder`,
`classification_report`, `RandomizedSearchCV`, `StratifiedKFold`.
● `xgboost>=2.0` cho các mô hình XGBoost đa lớp (`xgboost.XGBClassifier`).
● `joblib` để lưu mô hình/encoder ra đĩa.
● Các thư viện `matplotlib`, `seaborn`, `jupyterlab` phục vụ visualization/thí nghiệm bổ sung.
Phần cứng: phát triển và chạy thử trên máy Apple M1 (8 nhân CPU, RAM 8 GB). Tập dữ liệu
nhỏ nên không yêu cầu GPU; XGBoost sử dụng `tree_method="hist"` để tận dụng CPU hiệu quả.
6.2. Siêu tham số (Hyperparameters)
6.2.1. XGBoost (baseline train scripts) 13 Tầng Tham số Giá trị
Continental (`scripts/train_continental_xgb.py`) `objective` `multi:softprob` `n_estimators` 200 `max_depth` 4 `learning_rate` 0.1 `subsample` 0.9 `colsample_bytree` 0.9 `eval_metric` `mlogloss` `tree_method` `hist` `random_state` 42
East Asia (`scripts/train_eastasian_xgb.py`)
Các giá trị khởi đầu giống tầng châu lục
6.2.2. XGBoost (hyperparameter tuning cho Đông Á)
Quá trình tinh chỉnh siêu tham số được thực hiện bằng RandomizedSearchCV kết hợp
StratifiedKFold 5 folds và tiêu chí đánh giá macro-F1. Không gian tìm kiếm:
- `n_estimators`: {150, 200, 300, 400, 500}
- `max_depth`: {2, 3, 4, 5, 6}
- `learning_rate`: {0.02, 0.05, 0.08, 0.1, 0.15}
- `subsample`: {0.7, 0.8, 0.9, 1.0}
- `colsample_bytree`: {0.7, 0.8, 0.9, 1.0}
- `min_child_weight`: {1, 2, 3, 5}
- `gamma`: {0, 0.1, 0.3, 0.5}
- `reg_lambda`: {0.5, 1.0, 1.5, 2.0}
Kết quả tốt nhất được fit lại trên toàn bộ tập East Asia để lưu `eastasia_xgb.pkl`.
6.2.3. Generative Bayesian Model 14
Siêu tham số chính: `smoothing_alpha = 1.0` (Beta prior uniform) áp dụng cho cả tầng châu
lục và Đông Á (`scripts/train_generative_bga.py`).
Priors cho từng lớp lấy theo tần suất mẫu thực tế (empirical). Trong quá trình inference có thể
điều chỉnh `threshold` của `predict_with_uncertainty` (mặc định 0.7) để cân bằng giữa độ
chính xác và tỷ lệ trả về `UNKNOWN`. 15 Phần 7.
Kết quả thực nghiệm
7.1. Kết quả thực nghiệm cho bài toán phân loại cấp Châu lục
Bảng 1. Bảng kết quả thực nghiệm cho bài toán phân loại cấp Châu lục
method accuracy mcc F1-Score AUC
AFR AMR EAS EUR SAS AFR AMR EAS EUR SAS XGBoost 0,97 0,96 0,99 0,88 1,00
0,96 0,98 1,00 0,99 1,00 1,00 1,00 GenerativeB GA 0,97 0,96 0,99 0,88 0,99
0,96 0,98 1,00 0,98 1,00 1,00 1,00
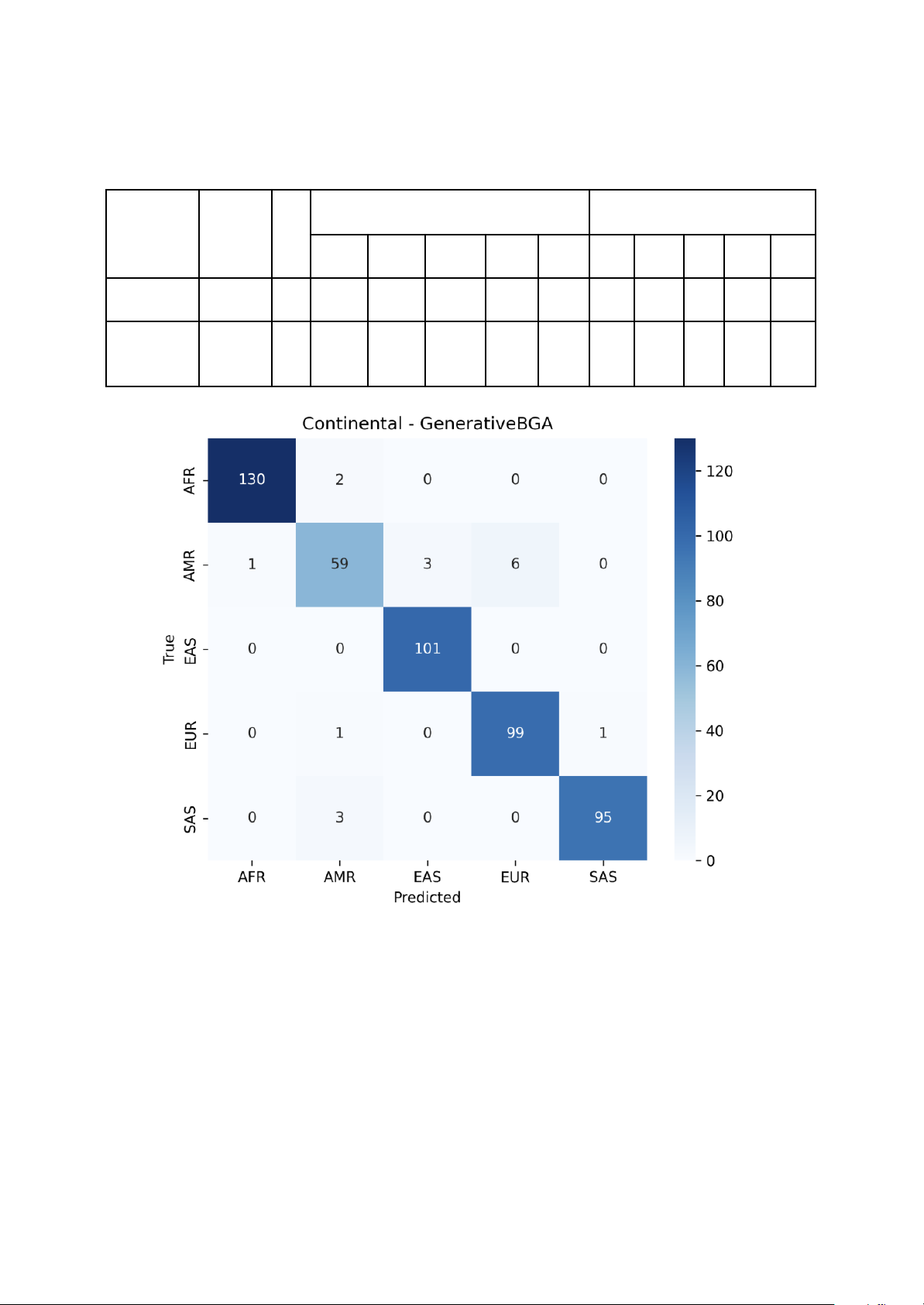
Hình 1: Confusion matrix cho mô hình Bayes trong bài toán phân loại cấp châu lục 16
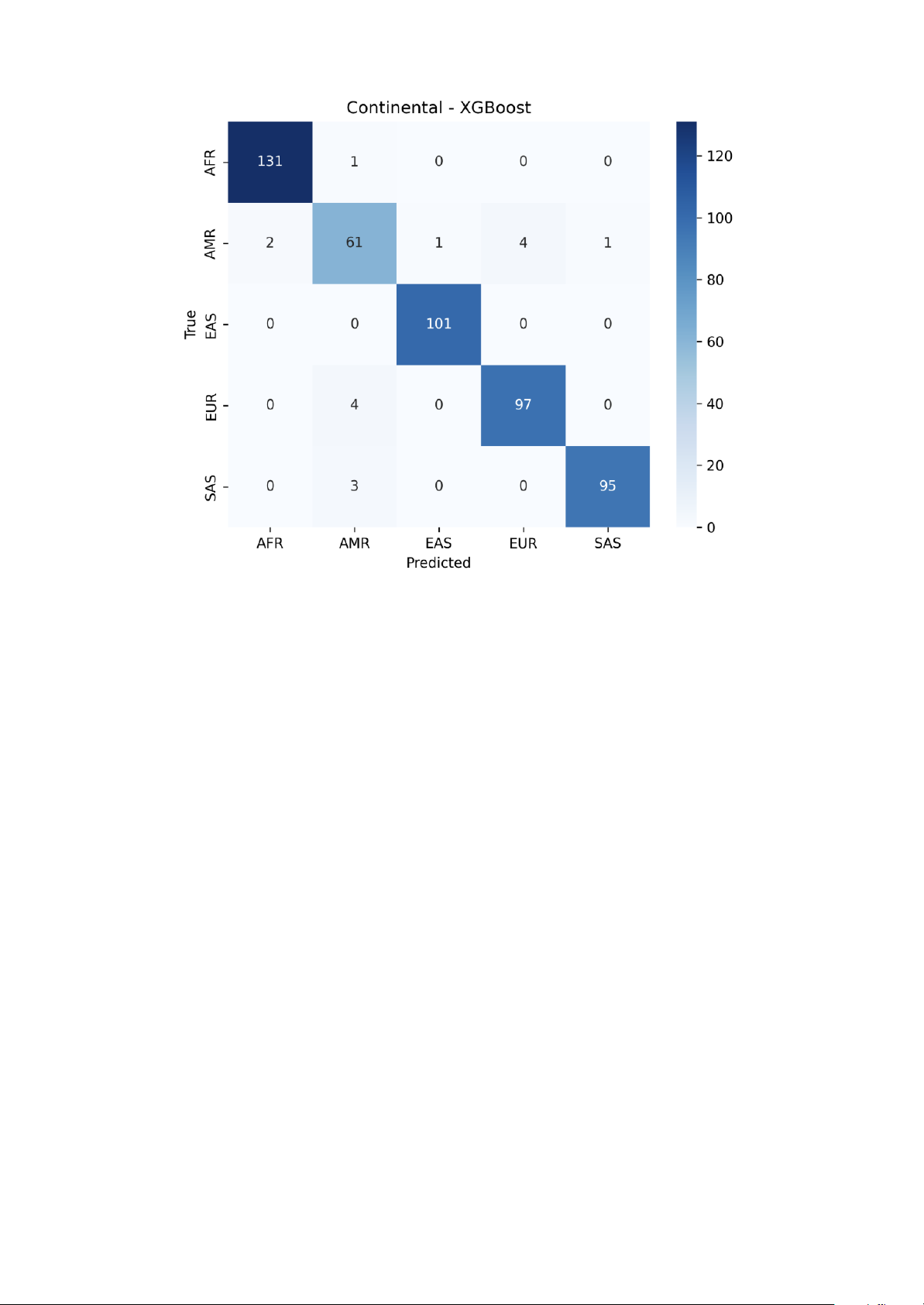
Hình 2: Confusion matrix cho mô hình XGBoost trong bài toán phân loại cấp châu lục
Ở tầng phân loại cấp châu lục, cả hai mô hình XGBoost và Generative Bayesian Model đều
cho thấy hiệu năng phân loại xuất sắc với độ chính xác (Accuracy) xấp xỉ 97%. Điều này
khẳng định 24 AISNP được chọn lọc có khả năng phân biệt rất mạnh mẽ giữa các nhóm tổ
tiên chính trên thế giới.
Phân tích hiệu năng tổng quát:
● XGBoost (Baseline): Đạt độ chính xác 96.81% và Macro-F1 score là 96.15%. Mô
hình thể hiện sự ổn định cao trên hầu hết các lớp.
● Generative Bayesian Model: Đạt độ chính xác 96.61% và Macro-F1 score là
95.90%. Kết quả này cho thấy dù không sử dụng quá trình tối ưu hóa gradient phức
tạp, mô hình Bayes vẫn nắm bắt rất tốt phân phối allele của các quần thể lớn.
Hiệu năng chi tiết trên từng nhóm quần thể: Dựa trên báo cáo phân loại (Classification
Report), ta có các nhận xét sau:
● Nhóm AFR (Châu Phi), EAS (Đông Á) và SAS (Nam Á): Cả hai mô hình đều đạt
điểm F1-score gần như tuyệt đối (từ 0.98 đến 1.00). Đặc biệt, nhóm EAS đạt Recall
100% trong cả hai mô hình, cho thấy các SNP lựa chọn cực kỳ đặc hiệu cho người Đông Á.
● Nhóm EUR (Châu Âu): Đạt F1-score ổn định ở mức 0.96.
● Nhóm AMR (Châu Mỹ): Đây là nhóm có hiệu năng thấp nhất (F1-score khoảng
0.88). Nguyên nhân chủ yếu do tính chất hỗn huyết (admixture) lịch sử của quần thể
Châu Mỹ, khiến dữ liệu genotype của nhóm này có sự giao thoa đáng kể với nhóm Châu Âu. 17
Phân tích ma trận nhầm lẫn (Confusion Matrix): Ma trận nhầm lẫn từ hình 1 và hình 2 cho thấy:
● Sai số chủ yếu tập trung vào việc nhầm lẫn giữa AMR và EUR. Cụ thể, mô hình
XGBoost nhầm 4 mẫu AMR sang EUR và ngược lại.
● Các nhóm AFR, EAS hầu như không bị nhầm lẫn với các nhóm khác, minh chứng cho
sự biệt hóa di truyền rõ rệt của các quần thể này đối với bộ SNP đang xét.
Nhìn chung, ở cấp độ châu lục, sự chênh lệch giữa mô hình phân biệt (XGBoost) và mô hình
sinh (Bayes) là không đáng kể (chỉ khoảng 0.2%). Điều này cho thấy với tập đặc trưng mạnh,
việc lựa chọn mô hình không quan trọng bằng chất lượng của các chỉ thị thông tin nguồn gốc (AIMs).
7.2. Kết quả thực nghiệm cho bài toán phân loại cấp Đông Á
Bảng 2. Bảng kết quả thực nghiệm cho bài toán phân loại cấp Đông Á
method accuracy mcc F1- Score AUC
CDX CHB CHS JPT KHV CDX CHB CHS JPT KHV XGBoost 0,69
0,62 0,68 0,61 0,63 0,98 0,56 0,92 0,90 0,83 1,00 0,87 Generative BGA 0,76
0,71 0,73 0,78 0,70 1,00 0,57 0,93 0,96 0,92 1,00 0,88
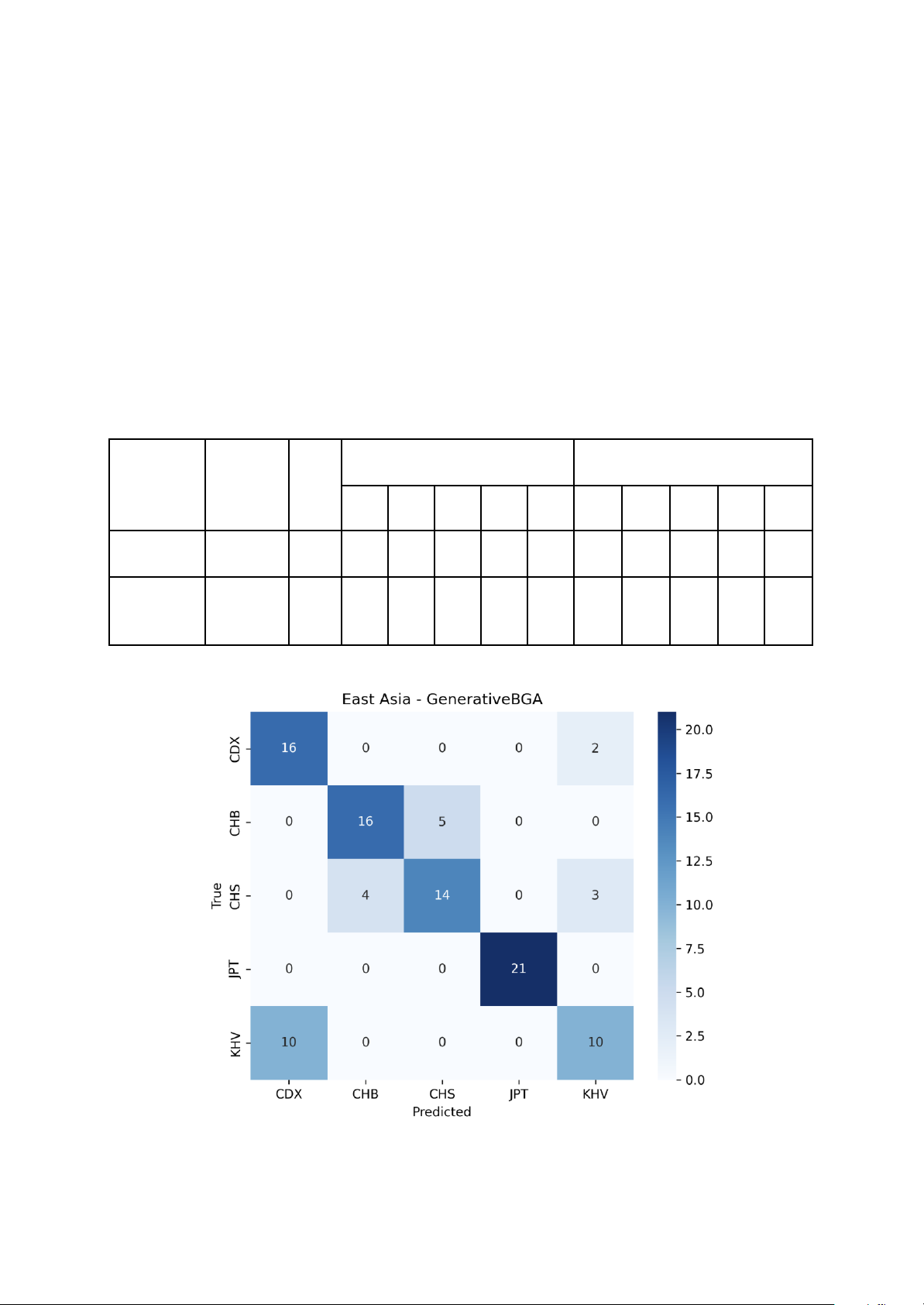
Hình 3: Confusion matrix cho mô hình Bayes trong bài toán phân loại cấp Đông Á 18
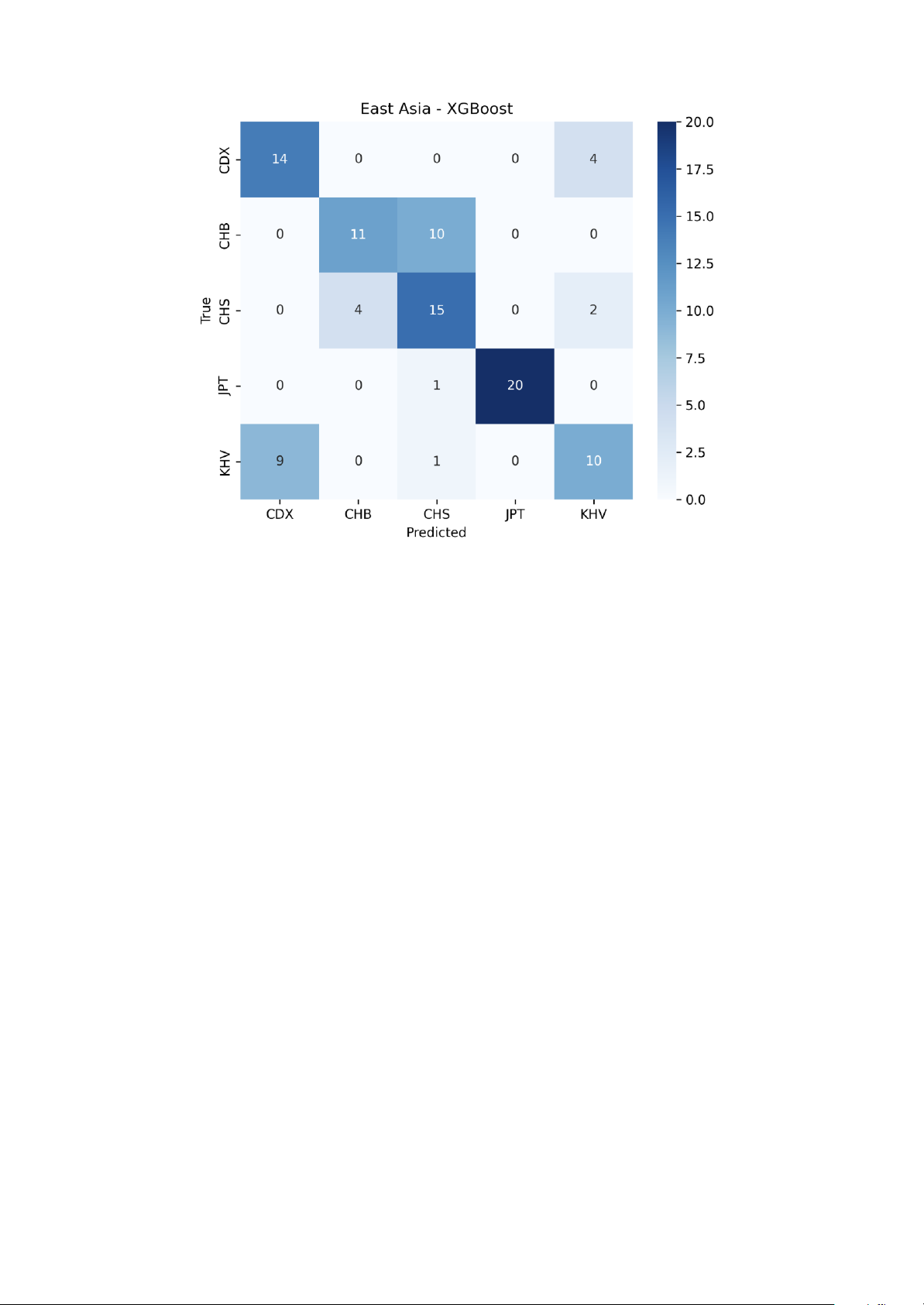
Hình 4: Confusion matrix cho mô hình XGBoost trong bài toán phân loại cấp Đông Á
Khác với cấp châu lục, bài toán phân loại chi tiết các quần thể con trong khu vực Đông Á
(EAS) cho thấy độ khó cao hơn hẳn do sự tương đồng lớn về kiểu gen giữa các nhóm dân cư lân cận.
Phân tích hiệu năng tổng quát:
● XGBoost (Baseline): Đạt độ chính xác 69.31% và Macro-F1 là 69.00%.
● Generative Bayesian Model: Đạt độ chính xác vượt trội hơn với 76.24% và Macro-F1 là 75.58%.
Kết quả này cho thấy trong bài toán phân loại chi tiết với kích thước mẫu nhỏ (504 mẫu), mô
hình sinh (Generative) với khả năng mô hình hóa trực tiếp tần suất allele tỏ ra hiệu quả hơn
các mô hình phân biệt dựa trên cây (Discriminative) như XGBoost.
Hiệu năng chi tiết trên từng nhóm quần thể:
● Nhóm JPT (Nhật Bản): Đạt kết quả gần như tuyệt đối trong cả hai mô hình (F1-score
từ 0.98 đến 1.00). Điều này cho thấy người Nhật Bản có sự biệt hóa di truyền rõ ràng
so với các quần thể lục địa Đông Á khác, dễ dàng nhận diện qua các AISNP.
● Nhóm CHB (Hán Bắc Kinh) và CHS (Hán Nam Trung Quốc): Đây là hai nhóm dễ
nhầm lẫn nhất. Ở mô hình XGBoost, Recall của CHB chỉ đạt 52.38%, nhiều mẫu bị
nhầm sang CHS. Điều này phản ánh sự giao thoa di truyền mạnh mẽ giữa các nhóm người Hán.
● Nhóm KHV (Kinh tại Việt Nam): Đạt F1-score khá thấp (0.55 - 0.57). Ma trận nhầm
lẫn cho thấy một lượng lớn mẫu KHV bị nhầm sang CDX (người Dai tại Trung Quốc)
và CHS, do các quần thể này có sự gần gũi về địa lý và lịch sử di cư. 19
Phân tích ma trận nhầm lẫn (Confusion Matrix): Dựa trên Hình 3 và Hình 4, ta nhận thấy:
● Sự nhầm lẫn mang tính hệ thống giữa các quần thể lục địa (CDX, CHB, CHS, KHV)
là thách thức lớn nhất.
● Mô hình Bayes xử lý tốt hơn việc phân tách CHB và CHS so với XGBoost, giúp nâng
tổng độ chính xác của toàn hệ thống lên thêm khoảng 7%.
Tóm lại, kết quả ở tầng 2 khẳng định rằng việc phân loại nguồn gốc sâu trong cùng một khu
vực địa lý đòi hỏi các phương pháp tiếp cận chuyên sâu hơn. Mô hình Bayes cho thấy ưu thế
rõ rệt khi làm việc với dữ liệu di truyền có độ tương đồng cao và kích thước mẫu hạn chế. 20



