














Preview text:
DỰ ĐOÁN NGUỒN GỐC TỔ TIÊN
ĐỊA LÝ-SINH HỌC (BGA) SỬ DỤNG DỮ LIỆU DNA
Học phần: Nhập môn học máy và khai phá dữ liệu
GVHD: PGS.TS Lê Đức Hậu, TS. Nguyễn Kiêm Hiếu Nhóm 8 Giới thiệu thành viên Trần Quí Đôn Nguyễn Minh Đức Phạm Hoàng Tiến Nguyễn Quốc Khánh Nguyễn Đình Duy 20235039 20235043 20230071 20235118 20235064 Giới thiệu bài toán Giới thiệu bài toán
Bài toán phân tích kiểu hình DNA pháp y cho nguồn gốc địa lý sinh học
(Biogeography Ancestry Prediction - BGA) là bài toán phân loại dữ liệu,
liên quan đến việc phân tích DNA thu được từ hiện trường vụ án.
Kết quả bài toán sẽ giúp cơ quan thực thi pháp luật xây dựng hoặc thu hẹp
phạm vi nghi phạm bằng cách cung cấp mô tả sinh học về nguồn gốc tổ
tiên của nghi phạm, đặc biệt hữu ích trong các cuộc điều tra khi không tìm
thấy kết quả khớp DNA trực tiếp.
Mục tiêu: phân loại nguồn gốc địa lý sinh học (BGA) sử dụng các Ancestry
Informative Single Nucleotide Polymorphisms (AISNP) thông qua hai tầng
phân loại chính: phân loại châu lục (continental) và phân loại quần thể Đông
Á chi tiết. Từ đó so sánh với các mô hình trước đó. Dữ liệu bài toán Dữ liệu bài toán Nguồn dữ liệu
Danh sách AISNP trích xuất từ Phụ lục
Supplementary Material 1 của bài báo
Systematic analyses of AISNPs screening and
classification algorithms based on genome-wide
data for forensic biogeographic ancestry inference
đăng trên tạp chí Forensic Science International: Genetics (2024)
Dữ liệu genotype được lấy từ 1000 Genomes
Project Phase 3, bao gồm 2.504 cá thể người
trong 5 châu lục và 26 quần thể Dữ liệu bài toán Mô tả dữ liệu
Dataset được thiết kế theo hai tầng phân loại: từ cấp độ châu lục đến cấp độ quần thể khu vực.
Về số lượng mẫu: Ở tầng châu lục, dataset gồm toàn bộ 2.504 mẫu từ
1000 Genomes Project, phân làm 5 nhóm chính là Châu Phi (AFR),
Châu Mỹ (AMR), Đông Á (EAS), Châu Âu (EUR) và Nam Á (SAS). Cụ thể
tầng Đông Á được giới hạn trong 504 mẫu, bao gồm 5 quần thể con.
Về đặc trưng: dataset sử dụng tổng cộng 58 SNP gồm 24 SNP ở cấp
châu lục và 34 SNP ở nội bộ Đông Á.
Thu thập và tiền xử lý dữ liệu
Dữ liệu Genotype được lấy từ dự án 1000 Genomes Project (file .vcf),
được thu thập bằng các script bash, lọc lấy thông tin của 58 AISNP và
dữ liệu về nguồn gốc địa lý của 2504 cá thể
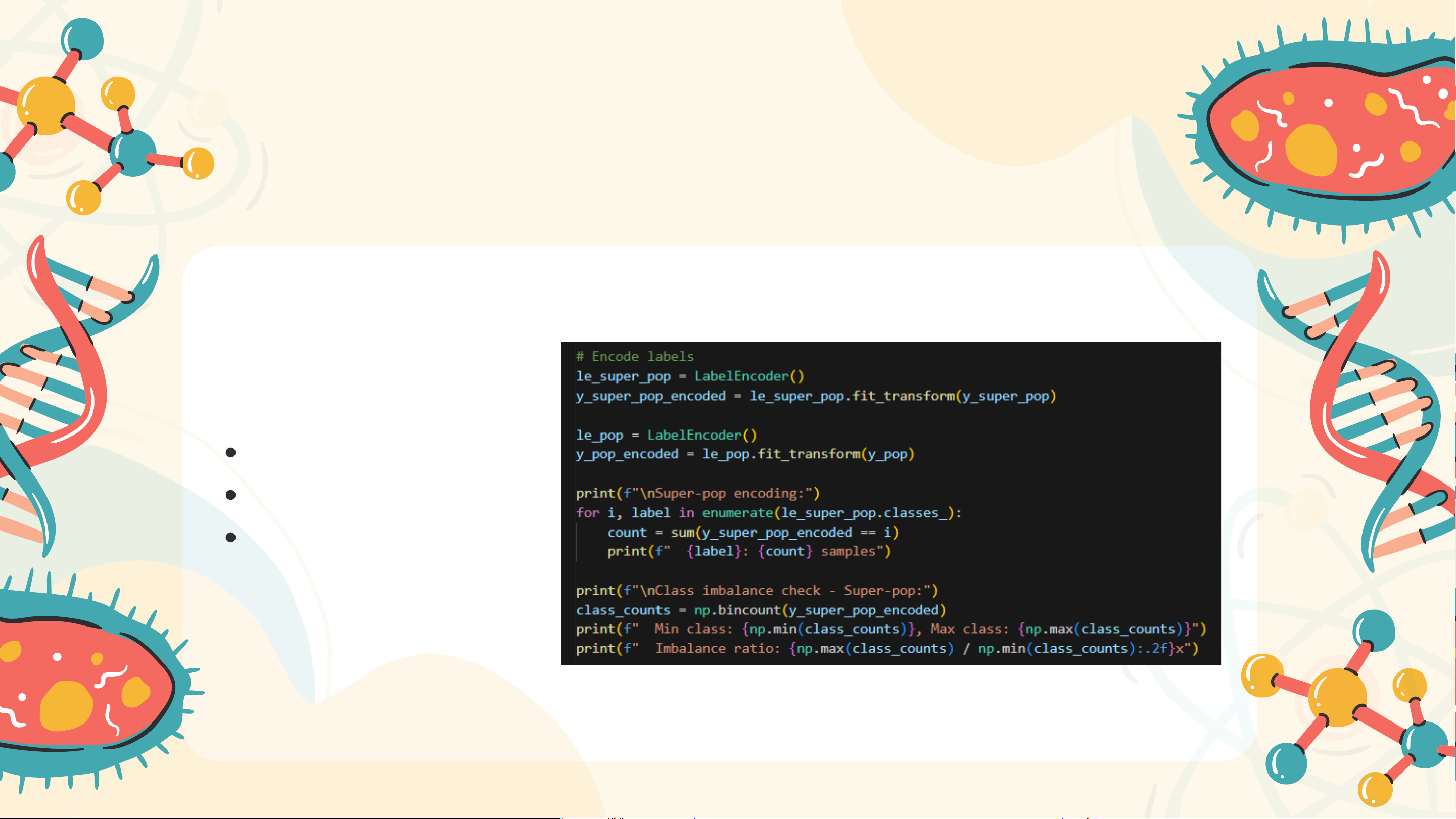
Dữ liệu genotype gốc ở dạng ký tự nucleotide (A, T, G, C) được chuyển
đổi sang dạng số thông qua bước encoding theo số lượng al ele minor: 0: đồng hợp al ele major 1: dị hợp 2: đồng hợp al ele minor Tiền xử lý dữ liệu
Sau khi dữ liệu được mã
hoá thực hiện các bước: Tách feature và label
Chuẩn hoá kiểu dữ liệu Mã hoá nhãn Mô hình đề xuất Mô hình đề xuất
Sử dụng mô hình phân loại hai tầng (Hierarchical classification)
Tầng 1 – Phân loại các châu lục
Bài toán: phân loại 5 lớp (AFR, EUR, EAS, SAS, AMR)
Dữ liệu: toàn bộ 2504 cá thể, sử dụng bộ 24 AISNP cho châu lục
Tầng 2: Phân loại chi tiết các quần thể trong khu vực Đông Á (EAS)
Thực hiện riêng cho khu vực Đông Á (EAS)
Huấn luyện dựa trên bộ 34 AISNP nội bộ cho khu vực Đông Á Thuật toán sử dụng XGBoost Generative Naïve Bayes Chiến lược huấn luyện
Chia dữ liệu: 80% Train – 20% Test
Đánh giá: Accuracy, Precision, Recal , F1-score Cơ sở lý thuyết Mô hình XGBoost
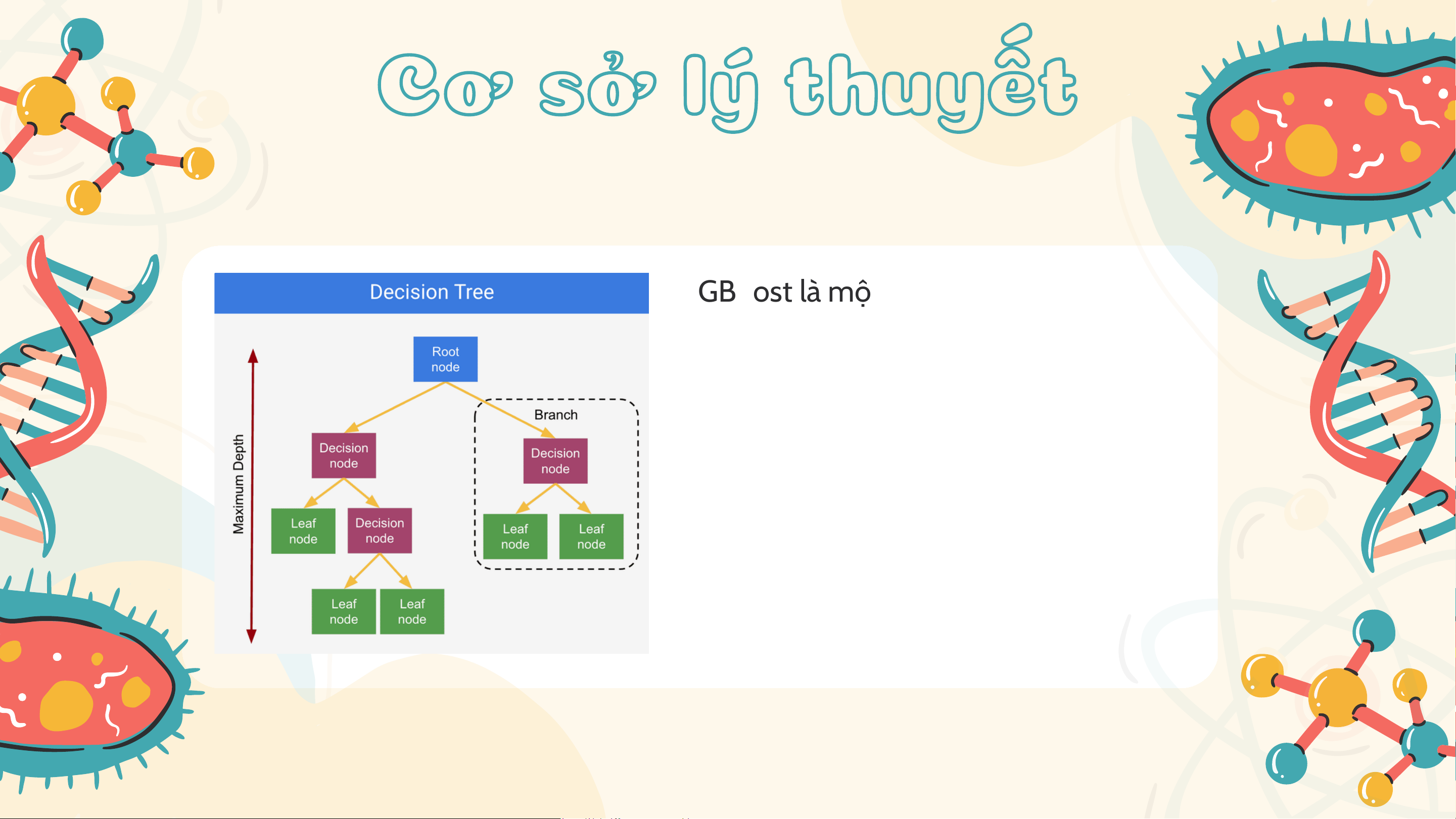
XGBoost là một thuật toán học máy dựa
trên cây quyết định (Decision Tree) kết hợp kỹ thuật Boosting Cơ sở lý thuyết Mô hình XGBoost
XGBoost phù hợp làm mô hình Baseline của bài toán, vì:
XGBoost xử lý rất tốt các đặc trưng thưa và không
cần chuẩn hóa dữ liệu đầu vào quá phức tạp.
Khả năng xếp hạng đặc trưng (Feature Importance) của XGBoost
XGBoost huấn luyện rất nhanh nhờ khả năng tính toán song song. Mô hình XGBoost Tham số khởi tạo Tham số Giá trị ` objective` ` multi:softprob` ` n_estimators` 200 ` max_depth` 4 ` learning_rate` 0,1 ` subsample` 0,9 ` colsample_bytree` 0,9 ` eval_metric` ` mlogloss` ` tree_method` ` hist` ` random_state` 42 Mô hình XGBoost
Quá trình tinh chỉnh siêu tham số được thực hiện bằng
RandomizedSearchCV kết hợp StratifiedKFold 5 folds và tiêu chí đánh giá macro-F1. Không gian tìm kiếm:
- `n_estimators`: {150, 200, 300, 400, 500}
- `max_depth`: {2, 3, 4, 5, 6}
- `learning_rate`: {0.02, 0.05, 0.08, 0.1, 0.15}
- `subsample`: {0.7, 0.8, 0.9, 1.0}
- `colsample_bytree`: {0.7, 0.8, 0.9, 1.0}
- `min_child_weight`: {1, 2, 3, 5} - `gamma`: {0, 0.1, 0.3, 0.5}
- `reg_lambda`: {0.5, 1.0, 1.5, 2.0} Cơ sở lý thuyết Mô hình Generative Bayesian
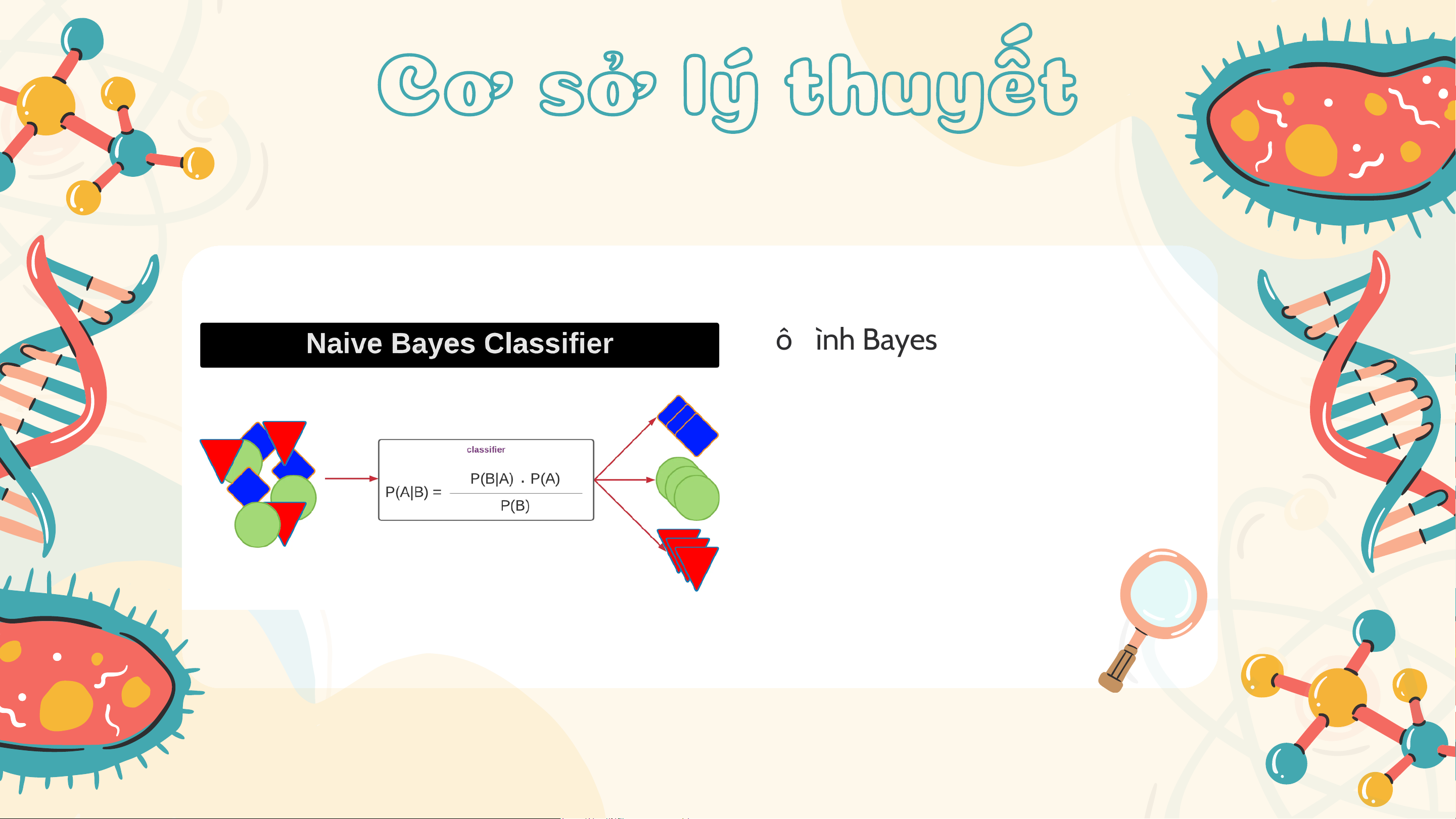
Mô hình Bayes sinh là mô hình
tập trung vào việc mô hình hóa
phân phối xác suất của dữ liệu
cho từng nhóm quần thể. VD Thuật toán STRUCTURE hoặc
các biến thể của Naive Bayes. Cơ sở lý thuyết Mô hình Generative Bayesian
Generative Bayesian Model phù hợp làm mô hình nâng cao của bài toán, vì:
Mô hình Bayes rất phù hợp với quy luật di truyền Mendel.
Mô hình Bayes cho phép tính toán tỷ lệ % tổ tiên Mô hình Generative Bayesian Thiết lập tham số:
Tham số chính: smoothing_alpha= 1.0
Ước lượng al ele frequency :
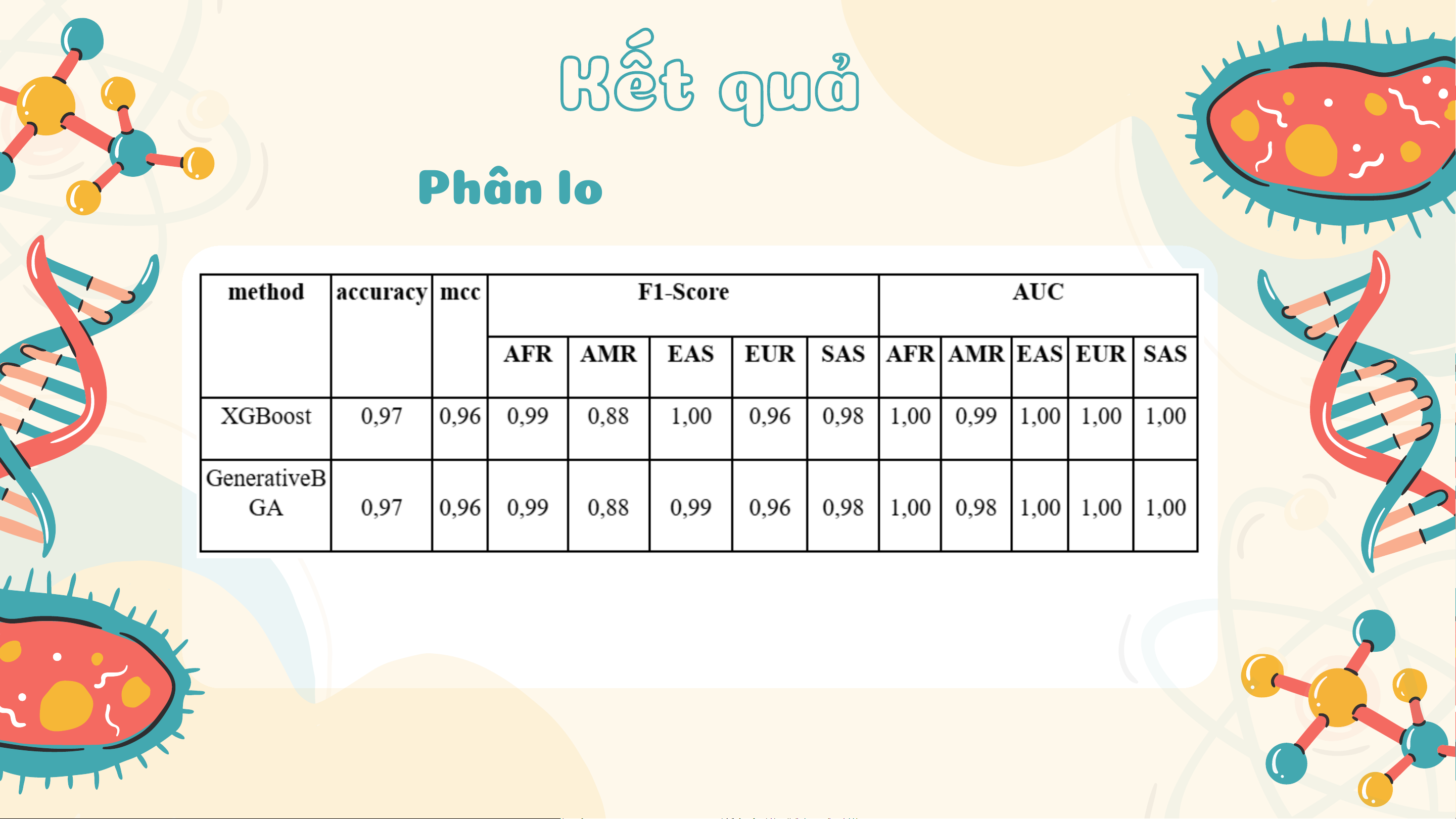
Nếu không có dữ liệu cho (k,j) → p = 0.5 (mặc định). Kết quả Kết quả Phân loại cấp Châu lục
Bảng kết quả thực nghiệm cho bài toán phân loại cấp Châu lục



